리보뉴클레오티드 환원효소
Ribonucleotide reductase| 리보뉴클레오시드-인산염 환원효소 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
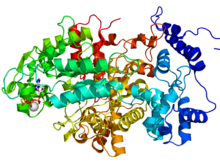
 리보뉴클레오시드-디프인산 환원효소 헤테오크타머, E.대장균 | |||||||||
| 식별자 | |||||||||
| EC 번호 | 1.17.4.1 | ||||||||
| CAS 번호. | 9047-64-7sy | ||||||||
| 데이터베이스 | |||||||||
| 인텐츠 | IntEnz 뷰 | ||||||||
| 브렌다 | 브렌다 입력 | ||||||||
| 엑스퍼시 | 나이스자이메 뷰 | ||||||||
| 케그 | KEG 입력 | ||||||||
| 메타사이크 | 대사통로 | ||||||||
| 프리암 | 프로필 | ||||||||
| PDB 구조 | RCSB PDB PDBe PDBsum | ||||||||
| 진 온톨로지 | 아미고 / 퀵고 | ||||||||
| |||||||||
리보뉴클레오티드 환원효소(RNR)는 리보뉴클레오시드 디포스포네이트 환원효소(rNDP)로도 알려져 있으며 리보뉴클레오티드에서 디옥시리보뉴클레오티드가 형성되는 것을 촉진하는 효소다.[1] 뉴클레오사이드 인산염 리보스 링의 2'히드록실 그룹을 제거하여 이러한 형성을 촉진한다. 이 감소는 디옥시리보뉴클레오티드를 생성한다.[2] 디옥시리보뉴클레오티드는 차례로 DNA의 합성에 사용된다. RNR에 의해 촉매된 반응은 모든 살아있는 유기체에서 엄격하게 보존된다.[3] 나아가 RNR은 세포분열과 DNA보수 과정에서 DNA 대 세포질량이 일정한 비율로 유지되도록 DNA 총합성률을 조절하는 데 중요한 역할을 한다.[4] RNR 효소의 다소 특이한 특징은 자유로운 급진적 작용 메커니즘을 통해 진행되는 반응을 촉진시킨다는 것이다.[5][6] RNR의 기판은 ADP, GDP, CDP, UDP이다. dTDP(deoxythymidine diphosphate)는 dTP(deoxymidine modine monophosphate)의 또 다른 효소(tymidylate kinase)에 의해 합성된다.
구조
리보뉴클레오티드 환원효과는 세 등급으로 나뉜다. 등급 I RNR 효소는 활성 이질체 테트라머를 형성하기 위해 연관되는 큰 알파 서브유닛과 작은 베타 서브유닛으로 구성된다. NDP를 2'-dNDP로 줄임으로써 효소는 DNA 합성 전구체로서 세포 증식에 필수적인 디옥시리보뉴클레오티드(dNTPs)의 디노보 합성을 촉진한다.[7] 클래스 II RNR는 아데노실코발라민에서 C-Co 결합의 균질 분할에 의해 5'데옥시아데노실 래디컬을 생성한다. 또한 클래스 III RNR에는 안정적인 글리실 라디컬이 포함되어 있다.[8]
인간은 1등급 RNR을 가지고 있다. 알파 서브 유닛은 RRM1 유전자에 의해 인코딩되며, 베타 서브 유닛의 두 개의 이소 형태가 있고, RRM2와 RRM2B 유전자에 의해 인코딩된다.
각 클래스 I 알파 모노머는 세 개의 도메인으로 구성된다.[9]
- 220 N-단자 잔류물로 구성된 헬리컬 영역 하나,
- 480개의 잔류물로 구성된 두 번째 대형 10 α/β 구조
- 70개의 잔류물로 구성된 세 번째 작은 5˚ α/β 구조.
Pfam에서 두 번째 도메인은 두 개의 별도 도메인으로 해석되었다.
- 짧은 전체 알파 N단자 영역,
- 그리고 더 긴 배럴 C-단자 영역.
|
|
|
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
클래스 I 베타 하위 유닛은 보통 디메탈 중심과 안정된 티로실 라디칼을 포함한다. 인간에게 있어서 베타 하위 단위는 디아이언 공동 요소에 의존한다. 대장균에서, 티로실 라디칼은 등급 I RNR2 하위 단위에 안정된 라디칼을 제공하는 위치 122 (Y122)에 위치한다.[13] A. aegytrighti에서 이 티로실 라디칼은 위치 184 (Y184)에 위치한다.[14] 티로실 라디칼은 티로실 라디칼의 안정화에 사용되는 철 중심에 가까운 소수성 환경의 단백질 내부에 깊이 매장되어 있다. 두 개의 μ-ox 연계 철의 구조는 철 결합 부위의 역할을 하는 리간드가 지배하고 있는데, 4개의 카르복실산염[아스파테이트 (D146), 글루탐산염 (E177, E240, E274)과 2개의 히스티딘 (H180, H277)이다.[14] RNR2의 C-단자와 RNR1의 C-단자 사이에 연관성이 발생한다.[9] 효소 활성은 RNR1 및 RNR2 서브유닛의 연결에 따라 달라진다. 활성 부위는 RNR1의 활성 디티올 그룹과 RNR2 서브 유닛의 이종 중심 및 티로실 라디컬로 구성된다.
아스파리트(D273), 트립토판(W48), 티로신(Y356)과 같은 RNR2의 다른 잔류물은 활성 사이트 티로실 래티컬을 더욱 안정화시켜 전자 전달을 허용한다.[9] 이러한 잔류물은 RNR2의 티로신(Y122)에서 RNR1의 시스테인(C439)으로 급진전자의 전달에 도움이 된다. 전자전달은 RNR2 타이로신(Y122)에서 시작하여 RNR2에서 트립토판(W48)으로 계속되는데, 트립토판은 RNR1 타이로신(Y731)에서 2.5나노미터 떨어져 있다. RNR2에서 RNR1로의 전자전송은 티로신(Y356에서 Y731까지)을 통해 발생하며 활성 부위의 티로신(Y730)에서 시스테인(C439)까지 계속된다.[15] RNR 1차 구조의 현장 주도적 변이는 위에서 인용한 모든 잔류물이 활성 사이트로의 자유 레디컬의 장거리 이동에 참여함을 나타낸다.[9]
In A. aegypti mosquitoes, RNR1 retains most of the crucial amino acid residues, including aspartate (D64) and valine (V292 or V284), that are necessary in allosteric regulation; proline (P210 and P610), leucine (L453 and L473), and methionine (M603) residues that are located in the hydrophobic active site; cysteine (C225, C436 and C451) residues that are involved in removal of a hydrogen atom and transfer of the radical electron at the active site; cysteine (C225 and C436), asparagine (N434), and glutamate (E441) residues that bind the ribonucleotide substrate; tyrosine (Y723 and Y743) residues that dictate the radical transfer; and cysteine (C838 and C841) residues that are used in the rege활성 사이트에서 dithiol 그룹 neration.[14]
함수
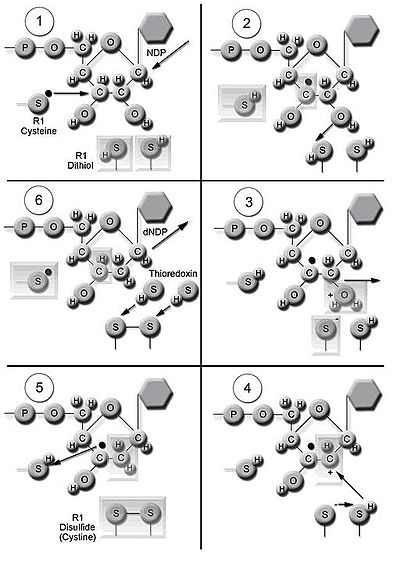
리보뉴클레오티드 환원효소(RNR)는 dNDP의 디노보합성을 촉진한다.[16] 리보뉴클레오시드 5'-디프인산염(NDP)의 강직투석에는 리보스 5-인산염의 2'-인산염을 감소시켜 2'-데옥시리보뉴클레오시드 5'-인산염(DDP)을 형성한다. 이 감소는 자유 급진주의자의 발생과 함께 시작된다. RNR은 단 한 번의 감소 후에 티오레독신 단백질의 디티올 그룹에서 기증된 전자를 요구한다. 티오레독신 재생은 니코틴아미드 아데닌 디뉴클레오티드 인산염(NADPH)이 티오레독신 이황화군 감소에 사용되는 수소원자 2개를 공급할 때 발생한다.
RNR의 세 종류는 NDP의 감소를 위한 유사한 메커니즘을 가지고 있지만, 자유 급진주의, 금속단백질 구조의 특정 금속, 전자 기증자를 생성하는 영역에서는 다르다. 모든 수업은 프리라디칼 케미를 사용한다.[9] 제1급 환원제는 철을 철로 변환하여 무타이로시릴 레디컬을 생성하기 위해 철로 된 철의 중심을 사용한다. NDP 기판 감소는 유산소 조건에서 발생한다. 등급Ⅰ 환원제는 규제의 차이로 인해 IA와 IB로 나뉜다. 클래스 IA 환원제는 진핵생물, 에우박테리아, 박테리오파지, 바이러스에 분포한다. 클래스 IB 환원제는 eubacteria에서 발견된다. 등급 IB 환원제도 이핵망간 중심 안정화에 따라 생성된 레디컬을 사용할 수 있다. 등급2 환원제는 코발라민(coenzyme B12)에서 5'-deoxyadenosyl rigical을 생성하며 등급1 및 등급3 환원제보다 구조가 단순하다. NDP 또는 리보뉴클레오티드 5'-삼인산염(NTP)의 감소는 에어로빅 또는 혐기성 조건에서 발생한다. Class II 환원제는 고고학, 에우박테리아, 박테리오파지에 분포한다. 등급3 환원제는 S-아데노실 메티오닌과 철 황 센터의 도움을 받아 생성된 글리신 라디칼을 사용한다. NTP의 감소는 혐기성 조건에 한정된다. 3급 환원제는 고고학, 에우박테리아, 박테리오파지에 분포한다.[9][14] 유기체는 한 종류의 효소를 갖는 것에 국한되지 않는다. 예를 들어 대장균은 1등급과 3등급 RNR을 모두 가지고 있다.
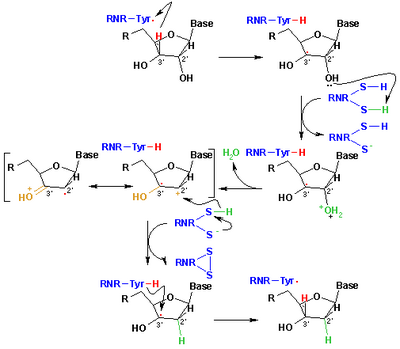
촉매 환원 메커니즘
디옥시리보뉴클레오티드에 대한 리보뉴클레오티드의 감소를 위해 현재 수용되고 있는 메커니즘이 다음과 같은 계획에 설명되어 있다. 첫 번째 단계는 급진적인 Cys439에 의한 기질 1의 3'-H 추상화를 포함한다. 그 후, 그 반응은 Cys225와 Glu441에 의해 촉매된 리보뉴클레오타이드의 탄소 C-2'에서 물 분자 1개를 제거하는 것을 포함한다. 세 번째 단계에서는 이전의 양성자가 Cys462에서 Cys225로 이전한 후 2'Kyel riginic 3의 Cys225에서 탄소 C-2'로 수소 원자 이전이 있다. 이 단계가 끝나면 급진적 음이온성 이황화 교량 및 폐쇄 쉘 케톤 중간 4를 얻는다. 이 중간은 효소 돌연변이와 상호작용하는 자연 기질뿐만[17] 아니라 여러 2'대체 기질 아날로그의 변환 과정에서 확인되었다. 다음 단계는 음이온 이황화 교량의 산화인데, 기질 감소가 동반되어 5가 생성된다. 스핀 밀도는 유황 원자에서 기질 C-3' 원자로 이동하며, 양성자가 Glu441에서 탄소 C-3'로 동시에 전달된다. 마지막 단계는 첫 번째 단계의 역순으로, Cys439에서 C-3'로 수소가 전달되어 초기 급진성이 재생되어 최종 6이 된다.
R1 단백질의 전체 모델을 사용하는 이러한 메커니즘의 일부 단계의 이론적 모델은 Cerqueira 등이 수행한 연구에서 확인할 수 있다.[18][19]
규정
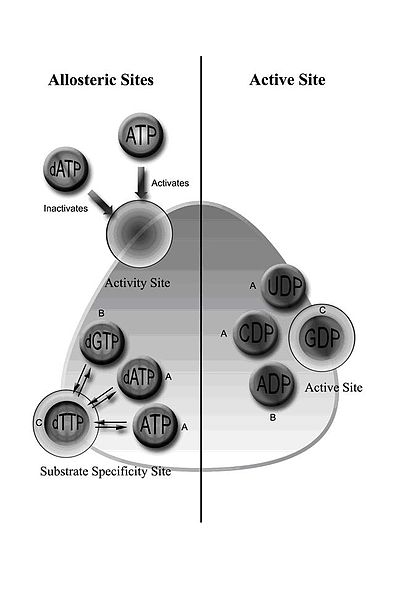
등급 I RNR은 RNR1과 RNR2 서브유닛으로 구성되며, RNR2 하위유닛은 이질성 테트라머를 형성하기 위해 연관될 수 있다.[5] RNR1은 기질 고유성 및 활성의 조절을 매개하는 두 개의 알로스테리학적 사이트를 포함한다.[11] 알로스테릭 구성에 따라 4개의 리보뉴클레오티드 중 1개가 활성 사이트에 바인딩된다.
RNR 규제는 dNTP의 균형 잡힌 양을 유지하도록 설계되었다. 이펙터 분자의 결합은 RNR 활동을 증가시키거나 감소시킨다. ATP가 알로스테릭 활동 부지에 결합하면 RNR을 활성화한다. 반대로 DATP가 이 사이트에 결합하면 RNR을 비활성화시킨다.[9] 알로스테리 메커니즘은 또한 활동을 제어하는 것 외에도 기질 특이성을 조절하고 효소가 DNA 합성을 위해 각 dNTP의 동일한 양을 생산하도록 한다.[9] In all classes, binding of ATP or dATP to the allosteric site induces reduction of cytidine 5’-diphosphate (CDP) and uridine 5’-diphosphate (UDP); 2’-deoxyguanosine 5’-triphosphate (dGTP) induces reduction of adenosine 5’-diphosphate (ADP); and 2’-deoxythymidine 5’-triphosphate (dTTP) induces reduction of guanosine 5’-diphosphate (GDP) (Figure 1).
등급 IB 환원제는 항원 활성 사이트에 필요한 약 50 N-단자 아미노산이 부족하기 때문에 dATP에 의해 억제되지 않는다.[20] 또한 리보뉴클레오티드 환원효소의 활동은 손상되지 않은 DNA의 합성이 디옥시리보뉴클레오티드의 균형 잡힌 풀에 의존하기 때문에 전사 및 전치 후 통제 하에 있는 것이 중요하다.[21] 등급 IA 환원제를 가진 진핵 세포는 축적되면서 dNTP의 합성을 끄기 위한 음의 제어 메커니즘을 가지고 있다. 이 메커니즘은 균형 잡힌 dNTP 풀의 변화가 DNA 손상과 세포 죽음으로 이어지기 때문에 dNTP의 과잉 생산에서 발생할 수 있는 독성 및 돌연변이 유발 효과로부터 세포를 보호한다.[22][23] dNTP의 과잉생산이나 불균형한 공급으로 인해 뉴클레오티드가 DNA로 잘못 결합될 수 있지만, dNTP의 공급은 DNA 수리를 가능하게 할 수 있다. p53R2는 리보뉴클레오티드 환원효소의 작은 단위로서 그러한 수리를 유도할 수 있다. 이 p53 유도 R2 호몰로겐 내의 변화는 미토콘드리아 DNA의 고갈을 야기할 수 있으며, 결과적으로 p53R2는 dNTP 공급의 주요 요인으로 작용한다.[24]
RNR는 알로스테릭 규제의 모르핀 모델을 사용할 수 있다.[25]
RNR1 및 RNR2 억제제
일반적으로 등급 I RNR 억제제는 효소의 합성을 차단하는 변환 억제제, 두 개의 RNR 서브유닛(R1 및 R2)의 연결을 막는 조광 억제제, 서브유닛 R1 및/또는 서브유닛 R2를 불활성화하는 촉매 억제제 등 3가지 주요 그룹으로 나눌 수 있다.[18]
등급 I RNR은 RNR2의 C-terminus와 유사한 펩타이드에 의해 억제될 수 있다. 이러한 펩타이드들은 RNR1과의 결합을 위해 RNR2와 경쟁할 수 있으며, 그 결과 RNR1은 RNR2와 함께 효소 활성 콤플렉스를 형성하지 않는다.[26][27] RNR2 단백질의 C-단자는 종마다 다르지만, RNR2는 종마다 RNR1과 상호작용할 수 있다.[28] 생쥐 RNR2 C-terminus를 대장균 RNR2 C-terminal(7 또는 33) 아미노산 잔류물로 교체할 때, 치메릭 RNR2 하위 유닛은 여전히 생쥐 RNR1 하위 유닛에 바인딩된다. 그러나, 그것들은 아마도 RNR2에서 RNR1 소부품으로의 자유 급진전자의 전달에 관련된 잔류물이 제거되었기 때문에 효소 활성도가 부족하다.[27]
작은 펩타이드들은 RNR2 서브유닛이 정상 RNR2 C-terminus와 유의미한 유사성을 공유할 때 RNR1과 결합하는 것을 특히 억제할 수 있다.[29] This inhibition RNR2 binding to RNR1 has been tested successfully in herpes simplex virus (HSV) RNR. When a 7 amino acid oligomer (GAVVNDL) truncated from the C-terminus of the RNR2 subunit was used in competition assays, it prevented the normal RNR2 from forming an enzymatically active complex with RNR1.[30] RNR2 C-terminus와 유사한 다른 작은 펩타이드 억제제들도 HSV RNR 효소 활성과 그에 따른 HSV 복제를 억제하는 데 성공적으로 사용되었다.[31] 쥐의 각막염과 각막신분자화(HSV 안구질환) 모델에서는 작은 RNR2 C단말기 아날로그 BILD 1263이 RNR을 억제하는 것으로 보고되어 이러한 질병을 예방하는 데 효과적이다.[32] 경우에 따라서는 작은 C단말기 아날로그로 치료해도 질병이 퍼지는 것을 막을 수는 없겠지만, 여전히 치유에 도움을 줄 수 있다. Acyclovir 내성 HSV(PAAr5)에서 소형 펩타이드 억제제 BILD 1633은 피하성 PAAr5 감염에 대해 BIL 1263보다 5~10배 더 강력한 것으로 보고되었다.[33] 결합 치료 접근법(BILD 1633과 acyclovir)은 생쥐의 국소 병변을 치료하는 데 더 효과적이다. 이러한 데이터는 RNR1과의 결합을 위해 RNR2와 경쟁하는 작은 펩타이드 억제제가 HSV의 확산을 방지하는 데 유용함을 시사한다.
갈륨은 활성 사이트에서 Fe를3+ 대체하여 RNR2를 억제한다. 말톨레이트 갈륨은 이 억제 활동을 이용하여 암, 감염 및 기타 질병을 치료하는 구강 생물학적으로 이용 가능한 갈륨의 형태다.[34]
히드록시루라와[35] 모텍사핀 가돌리늄 약물은 이 효소의 작용을 방해한다.[36]
참조
- ^ Elledge SJ, Zhou Z, Allen JB (March 1992). "Ribonucleotide reductase: regulation, regulation, regulation". Trends in Biochemical Sciences. 17 (3): 119–23. doi:10.1016/0968-0004(92)90249-9. PMID 1412696.
- ^ Sneeden JL, Loeb LA (September 2004). "Mutations in the R2 subunit of ribonucleotide reductase that confer resistance to hydroxyurea". The Journal of Biological Chemistry. 279 (39): 40723–8. doi:10.1074/jbc.M402699200. PMID 15262976.
- ^ Torrents E, Aloy P, Gibert I, Rodríguez-Trelles F (August 2002). "Ribonucleotide reductases: divergent evolution of an ancient enzyme". Journal of Molecular Evolution. 55 (2): 138–52. doi:10.1007/s00239-002-2311-7. PMID 12107591. S2CID 24603578.
- ^ Herrick J, Sclavi B (January 2007). "Ribonucleotide reductase and the regulation of DNA replication: an old story and an ancient heritage". Molecular Microbiology. 63 (1): 22–34. doi:10.1111/j.1365-2958.2006.05493.x. PMID 17229208. S2CID 9473163.
- ^ a b Eklund H, Eriksson M, Uhlin U, Nordlund P, Logan D (August 1997). "Ribonucleotide reductase—structural studies of a radical enzyme". Biological Chemistry. 378 (8): 821–5. doi:10.1515/bchm.1997.378.8.815. PMID 9377477.
- ^ Stubbe J, Riggs-Gelasco P (November 1998). "Harnessing free radicals: formation and function of the tyrosyl radical in ribonucleotide reductase". Trends in Biochemical Sciences. 23 (11): 438–43. doi:10.1016/S0968-0004(98)01296-1. PMID 9852763.
- ^ Fairman JW, Wijerathna SR, Ahmad MF, Xu H, Nakano R, Jha S, Prendergast J, Welin RM, Flodin S, Roos A, Nordlund P, Li Z, Walz T, Dealwis CG (March 2011). "Structural basis for allosteric regulation of human ribonucleotide reductase by nucleotide-induced oligomerization". Nature Structural & Molecular Biology. 18 (3): 316–22. doi:10.1038/nsmb.2007. PMC 3101628. PMID 21336276.
- ^ Larsson KM, Jordan A, Eliasson R, Reichard P, Logan DT, Nordlund P (November 2004). "Structural mechanism of allosteric substrate specificity regulation in a ribonucleotide reductase". Nature Structural & Molecular Biology. 11 (11): 1142–9. doi:10.1038/nsmb838. PMID 15475969. S2CID 1025702.
- ^ a b c d e f g h Jordan A, Reichard P (1998). "Ribonucleotide reductases". Annual Review of Biochemistry. 67 (1): 71–98. doi:10.1146/annurev.biochem.67.1.71. PMID 9759483.
- ^ a b PDB: 1PEU; Uppsten M, Färnegårdh M, Jordan A, Eliasson R, Eklund H, Uhlin U (June 2003). "Structure of the large subunit of class Ib ribonucleotide reductase from Salmonella typhimurium and its complexes with allosteric effectors". Journal of Molecular Biology. 330 (1): 87–97. doi:10.1016/S0022-2836(03)00538-2. PMID 12818204.
- ^ a b Uhlin U, Eklund H (August 1994). "Structure of ribonucleotide reductase protein R1". Nature. 370 (6490): 533–9. doi:10.1038/370533a0. PMID 8052308. S2CID 8940689.
- ^ Nordlund P, Eklund H (July 1993). "Structure and function of the Escherichia coli ribonucleotide reductase protein R2". Journal of Molecular Biology. 232 (1): 123–64. doi:10.1006/jmbi.1993.1374. PMID 8331655.
- ^ Högbom M, Andersson ME, Nordlund P (March 2001). "Crystal structures of oxidized dinuclear manganese centres in Mn-substituted class I ribonucleotide reductase from Escherichia coli: carboxylate shifts with implications for O2 activation and radical generation". Journal of Biological Inorganic Chemistry. 6 (3): 315–23. doi:10.1007/s007750000205. PMID 11315567. S2CID 20748553.
- ^ a b c d Pham DQ, Blachuta BJ, Nichol H, Winzerling JJ (September 2002). "Ribonucleotide reductase subunits from the yellow fever mosquito, Aedes aegypti: cloning and expression". Insect Biochemistry and Molecular Biology. 32 (9): 1037–44. doi:10.1016/S0965-1748(02)00041-3. PMID 12213240.
- ^ Chang MC, Yee CS, Stubbe J, Nocera DG (May 2004). "Turning on ribonucleotide reductase by light-initiated amino acid radical generation". Proceedings of the National Academy of Sciences of the United States of America. 101 (18): 6882–7. doi:10.1073/pnas.0401718101. PMC 406436. PMID 15123822.
- ^ Cox M, Nelson DR (2008). Lehninger Principles of Biochemistry. San Francisco: W. H. Freeman. ISBN 978-0-7167-7108-1.
- ^ Cerqueira NM, Fernandes PA, Eriksson LA, Ramos MJ (December 2004). "Ribonucleotide activation by enzyme ribonucleotide reductase: understanding the role of the enzyme". Journal of Computational Chemistry. 25 (16): 2031–7. doi:10.1002/jcc.20127. PMID 15481089. S2CID 19665974.
- ^ a b Cerqueira NM, Pereira S, Fernandes PA, Ramos MJ (2005). "Overview of ribonucleotide reductase inhibitors: an appealing target in anti-tumour therapy". Current Medicinal Chemistry. 12 (11): 1283–94. doi:10.2174/0929867054020981. PMID 15974997.
- ^ Cerqueira NM, Fernandes PA, Eriksson LA, Ramos MJ (March 2006). "Dehydration of ribonucleotides catalyzed by ribonucleotide reductase: the role of the enzyme". Biophysical Journal. 90 (6): 2109–19. doi:10.1529/biophysj.104.054627. PMC 1386789. PMID 16361339.
- ^ Eliasson R, Pontis E, Jordan A, Reichard P (October 1996). "Allosteric regulation of the third ribonucleotide reductase (NrdEF enzyme) from enterobacteriaceae". The Journal of Biological Chemistry. 271 (43): 26582–7. doi:10.1074/jbc.271.43.26582. PMID 8900130.
- ^ Thelander L (June 2007). "Ribonucleotide reductase and mitochondrial DNA synthesis". Nature Genetics. 39 (6): 703–4. doi:10.1038/ng0607-703. PMID 17534360. S2CID 22565931.
- ^ Kunz BA (1988). "Mutagenesis and deoxyribonucleotide pool imbalance". Mutation Research. 200 (1–2): 133–47. doi:10.1016/0027-5107(88)90076-0. PMID 3292903.
- ^ Meuth M (April 1989). "The molecular basis of mutations induced by deoxyribonucleoside triphosphate pool imbalances in mammalian cells". Experimental Cell Research. 181 (2): 305–16. doi:10.1016/0014-4827(89)90090-6. PMID 2647496.
- ^ Bourdon A, Minai L, Serre V, Jais JP, Sarzi E, Aubert S, Chrétien D, de Lonlay P, Paquis-Flucklinger V, Arakawa H, Nakamura Y, Munnich A, Rötig A (June 2007). "Mutation of RRM2B, encoding p53-controlled ribonucleotide reductase (p53R2), causes severe mitochondrial DNA depletion". Nature Genetics. 39 (6): 776–80. doi:10.1038/ng2040. PMID 17486094. S2CID 22103978.
- ^ Selwood T, Jaffe EK (March 2012). "Dynamic dissociating homo-oligomers and the control of protein function". Archives of Biochemistry and Biophysics. 519 (2): 131–43. doi:10.1016/j.abb.2011.11.020. PMC 3298769. PMID 22182754.
- ^ Climent I, Sjöberg BM, Huang CY (May 1991). "Carboxyl-terminal peptides as probes for Escherichia coli ribonucleotide reductase subunit interaction: kinetic analysis of inhibition studies". Biochemistry. 30 (21): 5164–71. doi:10.1021/bi00235a008. PMID 2036382.
- ^ a b Hamann CS, Lentainge S, Li LS, Salem JS, Yang FD, Cooperman BS (March 1998). "Chimeric small subunit inhibitors of mammalian ribonucleotide reductase: a dual function for the R2 C-terminus?". Protein Engineering. 11 (3): 219–24. doi:10.1093/protein/11.3.219. PMID 9613846.
- ^ Cosentino G, Lavallée P, Rakhit S, Plante R, Gaudette Y, Lawetz C, Whitehead PW, Duceppe JS, Lépine-Frenette C, Dansereau N (January 1991). "Specific inhibition of ribonucleotide reductases by peptides corresponding to the C-terminal of their second subunit". Biochemistry and Cell Biology. 69 (1): 79–83. doi:10.1139/o91-011. PMID 2043345.
- ^ Cooperman BS (2003). "Oligopeptide inhibition of class I ribonucleotide reductases". Biopolymers. 71 (2): 117–31. doi:10.1002/bip.10397. PMID 12767114. S2CID 25196379.
- ^ Filatov D, Ingemarson R, Gräslund A, Thelander L (August 1992). "The role of herpes simplex virus ribonucleotide reductase small subunit carboxyl terminus in subunit interaction and formation of iron-tyrosyl center structure". The Journal of Biological Chemistry. 267 (22): 15816–22. doi:10.1016/S0021-9258(19)49608-7. PMID 1322407.
- ^ Cohen EA, Gaudreau P, Brazeau P, Langelier Y (1986). "Specific inhibition of herpesvirus ribonucleotide reductase by a nonapeptide derived from the carboxy terminus of subunit 2". Nature. 321 (6068): 441–3. doi:10.1038/321441a0. PMID 3012360. S2CID 4238076.
- ^ Brandt CR, Spencer B, Imesch P, Garneau M, Déziel R (May 1996). "Evaluation of a peptidomimetic ribonucleotide reductase inhibitor with a murine model of herpes simplex virus type 1 ocular disease". Antimicrobial Agents and Chemotherapy. 40 (5): 1078–84. doi:10.1128/aac.40.5.1078. PMC 163269. PMID 8723444.
- ^ Duan J, Liuzzi M, Paris W, Lambert M, Lawetz C, Moss N, Jaramillo J, Gauthier J, Déziel R, Cordingley MG (July 1998). "Antiviral activity of a selective ribonucleotide reductase inhibitor against acyclovir-resistant herpes simplex virus type 1 in vivo". Antimicrobial Agents and Chemotherapy. 42 (7): 1629–35. doi:10.1128/aac.42.7.1629. PMC 105657. PMID 9660995.
- ^ Bernstein LR (December 1998). "Mechanisms of therapeutic activity for gallium" (PDF). Pharmacological Reviews. 50 (4): 665–82. PMID 9860806.
- ^ "Information on EC 1.17.4.1 – ribonucleoside-diphosphate reductase". Brenda. Retrieved 25 July 2015.
- ^ Hashemy SI, Ungerstedt JS, Zahedi Avval F, Holmgren A (April 2006). "Motexafin gadolinium, a tumor-selective drug targeting thioredoxin reductase and ribonucleotide reductase". The Journal of Biological Chemistry. 281 (16): 10691–7. doi:10.1074/jbc.M511373200. PMID 16481328.
외부 링크
- 미국 국립 의학 도서관의 리보뉴클레오티드+감소제(MeSH)
- 리보뉴클레오티드 환원효소 데이터베이스(RNRDB)