소아마비바이러스
Poliovirus| 소아마비바이러스 | |
|---|---|
 | |
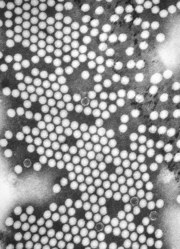
| 소아마비 바이러스 바이러스의 TEM 마이크로그래프.축척 막대(흰색): 50nm | |
 | |
| 제3형 소아마비 바이러스 캡시드, 단백질 사이드 체인 컬러 | |
| 바이러스 분류 | |
| (랭킹되지 않음): | 바이러스 |
| 영역: | 리보비리아 |
| 킹덤: | 오르토나비라과 |
| 망울: | 피수비리코타 |
| 클래스: | 피소니비리케테스 |
| 순서: | 피코르나비르목 |
| 패밀리: | 피코르나비르과 |
| 속: | 엔테로바이러스 |
| 종: | |
| 바이러스: | 소아마비바이러스 |
| 세로타입스 | |
소아마비의 원인 물질인 소아마비바이러스(poliomyelitis라고도 한다)는 피코르나비르과 계열의 엔테로바이러스 C종의 세로형이다.[1]소아마비 바이러스 세로타입은 유형 1, 2, 3이다.
소아마비 바이러스는 RNA 게놈과 단백질 캡시드로 구성되어 있다.게놈은 뉴클레오티드가 약 7500개인 단일 가닥의 양의 RNA(+ssRNA) 게놈이다.[2]바이러스 입자는 지름이 약 30nm이며, 이두면체 대칭이다.그것의 짧은 게놈과 그것을 캡슐화하는 RNA와 개발되지 않은 이코사면 단백질 외투의 단순한 구성 때문에, 폴리오바이러스는 가장 단순한 중요한 바이러스로 널리 간주되고 있다.[3]
폴리오바이러스는 1909년 칼 랜드스테이너와 에르윈 포퍼에 의해 처음으로 격리되었다.[4]이 바이러스의 구조는 1958년 로잘린드 프랭클린이 이끄는 버크벡 대학의 팀이 X선 회절법을 사용하여 처음으로 규명되었는데,[5][6] 소아마비 바이러스가 이두상 대칭을 이루고 있음을 보여준다.[7]
1981년, 소아마비 바이러스 게놈은 MIT의[8] 빈센트 라카니엘로와 데이비드 볼티모어에 의해 그리고 스토니 브룩 대학의 키타무라 나오미와 에카드 윔머에 의해 출판되었다.[9]
소아마비 바이러스의 3차원 구조는 1985년 제임스 호글이 스크립스 연구소의 X선 결정학을 이용해 결정했다.[10]
소아마비바이러스는 가장 성격이 좋은 바이러스 중 하나로 RNA 바이러스의 생물학을 이해하는 데 유용한 모델 시스템이 되었다.
복제 주기
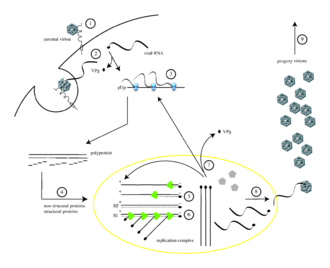
소아마비 바이러스는 세포 표면의 면역글로불린과 유사한 수용체인 CD155(일명 소아마비바이러스 수용체 또는 PVR이라고도 한다)[12][13]에 결합하여 인간 세포를 감염시킨다.[14]소아마비 바이러스와 CD155의 상호작용은 바이러스 입자에 필요한 바이러스 입자의 되돌릴 수 없는 순응적 변화를 촉진한다.[15][16]숙주 세포막에 부착된 후, 바이러스 핵산의 진입은 두 가지 방법 중 하나로, 즉 RNA가 숙주 세포질에 "주입"되는 혈장 막의 기공 형성을 통해 발생하거나, 수용체 매개 내세포 분열에 의한 바이러스 흡수를 통해 발생한다고 생각되었다.[17]최근의 실험 증거는 후자의 가설을 뒷받침하며 소아마비 바이러스가 CD155에 결합되어 내분비증에 의해 취해진다는 것을 시사한다.입자의 내실화 직후 바이러스 RNA가 방출된다.[18]
소아마비바이러스는 양성 변형 RNA 바이러스다.따라서 바이러스 입자 내에 밀폐된 게놈은 메신저 RNA로 사용할 수 있고 숙주세포에 의해 즉시 번역될 수 있다.침입 시, 바이러스는 세포의 번역기계를 강탈하여 바이러스 특이 단백질 생산을 위해 세포 단백질 합성을 억제한다.[19]숙주세포의 mRNA와는 달리 소아마비바이러스 RNA의 5' 끝부분은 700개 이상의 뉴클레오티드가 매우 길고 구조도 매우 높다.바이러스 게놈의 이 영역을 내부 리보솜 진입 사이트(IRES)라고 한다.이 지역은 많은 이차 구조와 3, 4개의 도메인으로 구성되어 있다.도메인 3은 두 개의 4방향 결합으로 연결된 다양한 안정적인 줄기 루프에 보존된 구조 모티브를 포함하고 있는 자체 접이식 RNA 소자다.IRES는 많은 도메인으로 구성되어 있기 때문에, 이들 도메인 자체는 리보솜을 납치하여 5' 끝마감 없이 수정된 번역에 기여하는 많은 루프들로 구성되어 있다.도메인 3의 상호작용 루프는 GNRA 4중주로 알려져 있다.GUAA 테트라루프의 아데노신 A180과 A181의 잔류물은 각각 수용체 C230/G242와 G231/C241의 염기쌍과의 비표준적 염기쌍 상호작용을 통해 수소 결합을 형성한다.[20]이 지역의 유전적 돌연변이는 바이러스성 단백질 생산을 막는다.[21][22][23]최초로 발견된 IRES는 소아마비바이러스 RNA에서 발견되었다.[24]
소아마비바이러스 mRNA는 하나의 긴 폴리펩타이드로 번역된다.이 폴리펩타이드제는 내부 단백질에 의해 약 10개의 개별 바이러스 단백질로 자동 압축된다.모든 갈라짐이 동일한 효율로 발생하는 것은 아니다.따라서 폴리펩타이드 갈라짐으로 생성되는 단백질의 양은 다양하다. 예를 들어, 3D의pol 적은 양이 캡시드 단백질인 VP1–4보다 생산된다.[25][26]이러한 개별 바이러스성 단백질은 다음과 같다.[3][27]

- 바이러스성 RNA 게놈의 복사를 여러 번 하는 기능이 있는 RNA 의존성 RNA 중합효소 3Dpol
- 2Apro 및 3Cpro/3CDpro, 바이러스성 폴리펩타이드 분쇄제
- VPG(3B)는 바이러스성 RNA를 결합하는 작은 단백질로 바이러스 양성 및 음성 스트랜드 RNA의 합성에 필요하다.
- 바이러스 복제에 필요한 단백질 복합체를 구성하는 2BC, 2B, 2C(ATPase),[28] 3AB, 3B 단백질.
- VP0, 바이러스 캡시드의 단백질인 VP2, VP4, VP1, VP3로 더 세분화된다.
단일 공정을 수반하는 번역, 전사, 게놈 복제 후에 (+)RNA의 합성이 실현된다.감염(+)RNA를 복제하려면 (-)RNA의 여러 사본을 필사한 다음 (+)RNA 합성을 위한 템플릿으로 사용해야 한다.템플릿 RNA와 다양한 길이의 몇 개의 성장하는 RNA로 구성된 RNA 분자의 결합인 복제 매개체(RIs)는 (-)RNA와 (+)RNA의 복제 콤플렉스에서 모두 볼 수 있다.각 음-스트랜드와 양-스트랜드 RNA의 합성을 위해 소아마비 바이러스의 VPG 단백질이 프라이머로 작용한다.소아마비바이러스의 RNA 의존성 RNA 중합효소는 음성-스트란드 항원성 RNA 합성을 위한 패턴으로 +ssRNA 게놈의 3′ 끝에서 폴리(A) 꼬리를 활용한 VPG 단백질에 2개의 우라실 뉴클레오티드(UUU)를 첨가한다.이 -ssRNA 합성을 시작하려면 VPg의 tyrosine hydroxyl이 필요하다.그러나 양의 스트랜드 RNA 합성을 시작하기 위해서는 CRE에 의존하는 VPg 요리디플레이션이 필요하다.즉, VPg가 한번 더 프라이머로 활용되지만 이번에는 시스 작용 복제 요소(CRE)를 템플릿으로 사용하여 두 개의 우리딘 삼인산(Uridine triphosphate)을 추가한다.[29][30]
소아마비 바이러스의 CRE는 61 nt로 구성된 미완성 염기성 줄기 및 최종 고리로 확인된다.CRE는 장내 기생충에서 발견된다.고도로 보존된 2차 RNA 구조 원소로 게놈의 다단백질 코딩 부위에 박혀 있다.이 복합체는 초기 위치에서 최소 3.7KB 떨어진 코딩 활동이 없는 게놈의 5' 영역으로 번역할 수 있다.이 과정은 활동에 부정적인 영향을 주지 않고 일어날 수 있다.CRE 복사본은 복제에 부정적인 영향을 미치지 않는다.CRE에서 일어나는 VPg의 iridlylation 공정은 RNA 결합 단백질인 3CD의pro 존재가 필요하다.그것은 직접적이고 구체적으로 CRE에 부착되어 있다.그 존재 때문에 VPg는 CRE를 제대로 묶을 수 있고 1차 생산은 문제없이 진행된다.[31]
일부 (+) RNA 분자는 추가 (-) RNA 합성을 위한 템플릿으로 사용되며, 일부는 mRNA로서 기능하며, 일부는 자손생물의 게놈으로 운명지어진다.[25]
새로운 바이러스 입자의 조합(즉, 유전체를 숙주 세포 외부에서 생존할 수 있는 프로캡시드로 포장)에는 각각 다음을 포함한다.[32]
- N 터미네이션과 VP4가 캡시드의 내부 표면을 형성하는 VP0, VP3, VP1 각각 5부씩 '펜타머'로 조립되고 12개의 펜타머가 프로캡시드를 형성한다.(캡시드의 외부 표면은 VP1, VP2, VP3, VP1 및 VP3의 C termini로 구성되며, 이 무렵 VP0의 60부는 VP4와 VP2로 분할된다.)
- 각 Procapsid는 바이러스 게놈의 복사본을 획득하고, VPG는 여전히 5의 끝에 부착된다.
완전히 조립된 소아마비 바이러스는 배양된 포유류 세포의 감염이 시작된 후 4시간에서 6시간 후에 용해함으로써[33] 숙주 세포의 경계를 떠난다.[34]세포로부터 바이러스 방출을 하는 메커니즘은 불분명하지만,[2] 각각의 죽어가는 세포는 최대 1만 개의 소아마비 바이러스를 방출할 수 있다.[34]
드레이크는 소아마비바이러스가 다중성 재활성화를 겪을 수 있다는 것을 증명했다.[35]즉, 소아마비바이러스가 자외선으로 조사되어 숙주세포의 다중 감염을 겪을 수 있게 되었을 때, 단일 감염에서 바이러스를 비활성화한 UV 선량에서도 생존 가능한 조생물이 형성될 수 있었다.소아마비 바이러스는 동일한 숙주 세포에 적어도 두 개의 바이러스 게놈들이 존재할 때 유전적 재조합을 겪을 수 있다.Kirkegaard와 볼티모어는[36] RdRP가 음극성 Strand 합성 동안 (+)ssRNA 템플릿 사이를 전환하는 복사 선택 메커니즘에 의해 RNA 의존성 RNA 중합효소(RdRP)가 재조합을 촉진한다는 증거를 제시했다.RNA 바이러스의 재결합은 게놈 손상을 복구하기 위한 적응적 메커니즘으로 보인다.[37][38]
원산지 및 세로타입
소아마비바이러스는 구조적으로 다른 인간 장로바이러스(콕사키예비루스, 에코비루스, 라이노비루스)와 유사하며, 면역글로불린과 같은 분자를 사용하여 숙주 세포를 인식하고 들어간다.[13]소아마비 바이러스의 RNA와 단백질 서열에 대한 유전학적 분석은 그것이 캡시드 내의 돌연변이를 통해 발생한 C-클러스터 Coxackie A 바이러스 조상으로부터 진화했을 수도 있음을 시사한다.[39]소아마비 바이러스의 구별되는 특징은 아마도 C-cluster Coxackie A 바이러스에 의해 사용되는 세포간 접착 분자-1(ICAM-1)에서 CD155로 세포 수용체 특이성이 변화하여 병원성 변화가 일어나며, 바이러스가 신경 조직을 감염시킬 수 있게 한 결과일 것이다.
바이러스 내 돌연변이 비율은 1.0 x 10−2 대체율/사이트/년, 비 동의어 대체율−4 3.0 x 10 대체율/사이트/년인 RNA 바이러스에서도 비교적 높다.[40]게놈 내의 염기분포는 무작위가 아니며, 아데노신이 5의 끝에서 예상보다 덜 흔하고 3의 끝에서 더 높다.[41]코돈 사용은 무작위로 이루어지는 것이 아니며 아데노신으로 끝나는 코돈은 선호되고 시토신이나 구아닌으로 끝나는 코돈은 피한다.코돈 사용은 세 가지 유전자형에 따라 다르며 선택보다는 돌연변이에 의해 구동되는 것으로 보인다.[42]
소아마비 바이러스인 PV-1, PV-2, PV-3의 세로형은 각각 약간 다른 캡시드 단백질을 가지고 있다.캡시드 단백질은 세포 수용체 특이성과 바이러스 항원성을 정의한다.PV-1은 자연에서 만나는 가장 흔한 형태지만 세 가지 형태 모두 극도로 전염성이 강하다.[4]2020년 3월 현재, 야생 PV-1은 파키스탄과 아프가니스탄의 지역에 고도로 국산화되어 있다.토종 전송 근절 인증은 야생 PV-2에 대해 2015년 9월, 1999년에 마지막으로 검출된 후,[43] 2012년에 마지막으로 검출된 후 2019년 10월에 이뤄졌다.[44]
각 세로타입의 특정 변종은 소아마비 예방 백신을 준비하는데 사용된다.비활성 소아마비 백신은 세 가지 야생적이고 치명적인 참조 변종인 마호니 또는 브루넨더(PV-1), MEF-1/랜싱(PV-2), 사우켓/레온(PV-3)의 포름틴 불활성화에 의해 준비된다.경구용 소아마비 백신은 세 가지 유형의 소아마비 바이러스 중 살아있는 감쇠(약화된) 변종을 포함한다.원숭이 콩팥 상피 세포에 바이러스 변종을 전달하면 바이러스 IRES에 돌연변이가 발생하며, 신경 조직을 감염시키는 바이러스의 능력을 저해(또는 약화)[34]한다.
소아마비루스는 이전에 피코르나비르과에 속하는 엔테로바이러스에 속하는 뚜렷한 종으로 분류되었다.2008년, 소아마비루스 종은 제거되었고 세로타입은 피코르나비르과의 엔테로바이러스 속 인간 엔테로바이러스 C(인체명 엔테로바이러스 C) 종에 할당되었다.엔토바이러스의 종류는 소아마비바이러스에서 (인간) 엔토바이러스 C로 바뀌었다.[45]
병생성
어떤 바이러스에 대한 감염의 주요 결정요인은 세포에 들어가 추가적인 감염 입자를 생성하는 능력이다.CD155의 존재는 소아마비 바이러스에 감염될 수 있는 동물과 조직을 정의하는 것으로 생각된다.CD155는 인간, 고등 영장류, 구세계 원숭이의 세포에서만 발견된다.그러나 소아마비바이러스는 엄격히 인간의 병원체로서 다른 종을 자연적으로 감염시키지 않는다(침팬지와 구세계원숭이는 실험적으로 감염될 수 있다).[46]
CD155 유전자는 양성의 선택을 받은 것으로 보인다.[47]이 단백질은 D1 도메인이 소아마비 바이러스 결합 사이트를 포함하는 여러 도메인을 가지고 있다.이 영역 내에서 37개의 아미노산이 바이러스의 결합을 담당한다.
소아마비 바이러스는 장내 바이러스다.염증은 분변-구강 경로를 통해 발생하는데, 이는 바이러스를 섭취하고 바이러스 복제가 소화관에서 발생한다는 것을 의미한다.[48]바이러스는 감염된 사람의 배설물에서 유출된다.95%의 경우 1차적이고 일시적인 바이레미아(혈류 내 바이러스)만 발생하며 소아마비바이러스 감염은 증상이 없다.약 5%의 경우, 갈색 지방, 망막 조직, 근육과 같은 다른 부위에서 바이러스가 퍼지고 복제된다.지속적인 바이러스 복제는 2차적인 빈혈증을 유발하고 발열, 두통, 인후염과 같은 경미한 증상의 발달을 이끈다.[49]마비성 폴리오밀염은 소아마비 바이러스 감염의 1% 미만에서 발생한다.마비성 질환은 바이러스가 중추신경계(CNS)에 들어가 척수, 뇌간, 운동피질 내 운동신경세포에서 복제해 운동신경세포가 선택적으로 파괴돼 일시적 또는 영구적 마비로 이어질 때 발생한다.이것은 아기들에게 매우 드문 일로, 아기들은 여전히 어머니로부터 얻은 항폴리오 바이러스 항체를 가지고 있다.[50]드물게 마비성 폴리오밀염은 호흡기 장애와 사망으로 이어진다.마비성 질환의 경우 근육통이나 경련이 일어나기 전 경련이 자주 나타난다.마비는 보통 회복되기 며칠 전부터 몇 주까지 지속된다.[51]
많은 점에서 신경학적 감염 단계는 정상적인 위장 감염의 우발적인 전환이라고 생각된다.[17]소아마비바이러스가 CNS에 들어가는 메커니즘은 잘 이해되지 않는다.그것의 입력을 설명하기 위해 세 개의 비합리적으로 배타적인 가설이 제안되었다.모든 이론은 일차적인 바이레미아를 필요로 한다.첫 번째 가설은 처녀자리들이 CD155와는 무관하게 혈액-뇌장벽을 통과함으로써 혈액으로부터 중추신경계로 직접 전달된다고 예측한다.[52]두 번째 가설은 처녀자리들이 역행하는 축수 이송을 통해 신경통로를 통해, 예를 들어 근육 조직과 같은 약혈에 씻겨진 말초 조직에서 척수로 옮겨진다는 것을 암시한다.[53][54][55]세 번째 가설은 바이러스가 감염된 단세포나 대식세포 등을 통해 CNS로 유입된다는 것이다.[11]
폴리오밀염은 중추신경계의 병이다.그러나 CD155는 대부분의 또는 모든 인간 세포의 표면에 존재하는 것으로 여겨진다.따라서 수용체 발현에서는 소아마비바이러스가 특정 조직을 우선적으로 감염시키는 이유를 설명하지 않는다.세포 감염 후 조직 열대가 결정된다는 의미다.최근 연구에서는 I형 인터페론 반응(특히 인터페론 알파와 베타형)이 소아마비 바이러스 복제를 지원하는 세포 유형을 정의하는 중요한 요소라고 제시하였다.[56](유전공학을 통해) CD155를 발현하지만 I형 인터페론 수용체가 부족한 생쥐에서 소아마비바이러스는 조직 유형의 확장된 레퍼토리에서 복제될 뿐만 아니라 이들 생쥐도 구강적으로 바이러스에 감염될 수 있다.[57]
면역체계 회피
소아마비 바이러스는 면역 체계를 피하기 위해 두 가지 주요 메커니즘을 사용한다.첫째, 위장의 고산성 상태를 견뎌낼 수 있어 바이러스가 숙주를 감염시키고 림프계를 통해 온몸으로 퍼질 수 있다.[3]둘째로, 그것은 매우 빠르게 복제될 수 있기 때문에, 바이러스는 면역 반응이 올라가기 전에 숙주 기관을 압도한다.[58]부착 단계에서 세부사항이 제시된 경우, 바이러스 표면에 협곡이 있는 소아마비바이러스는 협곡 베이스의 포켓에 바이러스 부착 사이트를 가지고 있다.협곡은 항체로 접근하기에 너무 좁기 때문에 바이러스 부착 부위는 숙주의 면역 감시로부터 보호되는 반면, 나머지 바이러스 표면은 숙주의 면역 반응을 피하기 위해 변이될 수 있다.[59]
소아마비 바이러스에 감염되거나 소아마비 백신을 접종함으로써 노출된 개인은 면역력을 발달시킨다.면역 개인에서 소아마비 바이러스에 대한 항체는 편도선과 위장관(특히 IgA 항체)에 존재하며 소아마비 바이러스 복제를 차단할 수 있다. 소아마비 바이러스에 대한 IgG와 IgM 항체는 중앙 신경계의 운동 신경세포로 바이러스가 전파되는 것을 막을 수 있다.[34]소아마비 바이러스의 한 세로타입에 감염되는 것은 다른 세로타입에 대한 면역력을 제공하지 않는다. 그러나 같은 개인 내의 두 번째 공격은 극히 드물다.[60]
PVR 유전자 변형 마우스
비록 인간이 소아마비 바이러스의 유일한 자연 숙주라고 알려져 있지만, 원숭이는 실험적으로 감염될 수 있고 그들은 오랫동안 소아마비 바이러스를 연구하는데 사용되어 왔다.1990-91년에, 폴리오멜리염의 작은 동물 모델이 두 개의 연구소에 의해 개발되었다.생쥐는 인간 수용체를 소아마비 바이러스(hPVR)에 표현하도록 설계되었다.[61][62]
일반 생쥐와 달리 유전자변형 소아마비바이러스 수용체(TgPvR) 생쥐는 정맥 또는 근육내 주입된 소아마비 바이러스에 취약하며, 척수나 뇌에 직접 주사하면 소아마비 바이러스에 감염되기 쉽다.[63]감염 시 TgPvR 생쥐는 사람과 원숭이의 폴리오밀염과 유사한 마비 징후를 보이며, 마비된 생쥐의 중추신경계는 사람과 원숭이의 중추신경계와 유사하게 조직화학적으로 유사하다.인간 소아마비 바이러스 감염의 이 마우스 모델은 소아마비 바이러스 생물학과 병원성을 이해하는 데 있어 귀중한 도구임이 입증되었다.[64]
TgPvR 생쥐의 세 가지 뚜렷한 유형이 잘 연구되었다.[65]
- TgPvR1 생쥐에서 인간 PVR을 인코딩하는 트랜스젠은 생쥐 4번 염색체에 통합되었다.이 쥐들은 트랜스젠의 가장 높은 수준과 소아마비 바이러스에 대한 가장 높은 민감도를 나타낸다.TgPvR1 생쥐는 경구 경로가 아니라 척추 내, 척추 내, 근육 내, 정맥 내 경로를 통해 소아마비 바이러스에 감염되기 쉽다.
- TgPvR21 생쥐는 13번 염색체에 인간 PVR을 통합했다.이러한 생쥐들은 추간내 경로를 통해 소아마비 바이러스 감염에 덜 취약하며, 아마도 그들이 hPvR의 감소된 수준을 표현하기 때문일 것이다.TgPvR21 생쥐는 뇌내 접종을 통해 소아마비 바이러스 감염에 취약한 것으로 나타났으며 점막 감염 모델로 유용할 수 있다.[66]
- TgPvR5 생쥐에서 인간의 트랜스젠은 12번 염색체에 위치한다.이러한 생쥐는 hPvR 발현 수준이 가장 낮으며 소아마비 바이러스 감염에 가장 취약하다.
최근 네 번째 TgPvR 마우스 모델이 개발되었다.이러한 "cPVR" 생쥐는 β-actin 프로모터에 의해 구동되는 hPvR cDNA를 운반하며, 척추 내, 근육 내 및 뇌 내 경로를 통해 소아마비 바이러스에 취약하다는 것이 입증되었다.게다가, 이 쥐들은 소아마비 예방 접종 후에 볼록한 형태의 소아마비를 개발할 수 있다.[66]
TgPvR 마우스의 개발은 경구 소아마비바이러스 백신(OPV) 생산에 지대한 영향을 미쳤다.이전에는 영장류만이 바이러스에 취약하기 때문에 원숭이를 사용하여 OPV의 안전성을 감시해야 했다.1999년 세계보건기구는 소아마비 바이러스 유형 3에 대한 백신의 효과를 평가하는 대안적 방법으로 TgPvR 마우스의 사용을 승인했다.2000년에 마우스 모델은 타입 1과 타입 2 소아마비 바이러스에 대한 백신 테스트를 위해 승인되었다.[67]
복제 및 합성
1981년 Racaniello와 볼티모어는 동물 RNA 바이러스의 첫 번째 감염성 클론인 소아마비 바이러스를 생성하기 위해 재조합 DNA 기술을 사용했다.소아마비 바이러스의 RNA 게놈을 인코딩하는 DNA가 배양된 포유류 세포에 도입되고 전염성 소아마비 바이러스가 생성되었다.[68]전염성 클론의 창조는 소아마비 바이러스 생물학의 이해를 촉진시켰고, 다른 많은 바이러스를 연구하는 데 사용되는 표준 기술이 되었다.
2002년 스토니브룩 대학의 에카드 윔머 그룹이 화학 코드에서 소아마비 바이러스를 합성하는 데 성공하여 세계 최초의 합성 바이러스를 생산하였다.[69]과학자들은 먼저 소아마비바이러스가 발표한 RNA 염기서열 7741 염기서열을 DNA 염기서열로 변환했는데, DNA는 합성하기가 더 쉬웠기 때문이다.이 DNA 서열의 짧은 파편들은 우편 주문으로 입수되어 조립되었다.완전한 바이러스 게놈은 유전자 합성 회사에 의해 조립되었다.19개의 표지가 합성된 DNA에 통합되어 자연적인 소아마비 바이러스와 구별될 수 있었다.효소는 DNA를 자연상태인 RNA로 되돌리기 위해 사용되었다.다른 효소들은 RNA를 폴리펩타이드로 변환하여 기능적인 바이러스 입자를 생성하는데 사용되었다.이 모든 힘든 과정은 2년이 걸렸다.새로 채굴된 합성 바이러스는 PVR 유전자 변형 생쥐에 주입되어 합성 버전이 질병을 일으킬 수 있는지 여부를 판별했다.합성 바이러스는 쥐에게 복제, 감염, 마비 또는 사망을 일으킬 수 있었다.그러나 합성 버전은 원래 바이러스보다 1,000배에서 1만배 정도 약했는데, 아마도 추가된 표식기 중 하나 때문일 것이다.[70]
치료법 수정
PVSRIPO라고 불리는 소아마비 바이러스의 수정은 암에 대한 가능한 치료법으로서 초기 임상 실험에서 시험되었다.[71][needs update]
참조
- ^ Ryan KJ, Ray CG, eds. (2004). Sherris Medical Microbiology (4th ed.). McGraw Hill. ISBN 978-0-8385-8529-0.[페이지 필요]
- ^ a b Hogle JM (2002). "Poliovirus cell entry: common structural themes in viral cell entry pathways". Annual Review of Microbiology. 56: 677–702. doi:10.1146/annurev.micro.56.012302.160757. PMC 1500891. PMID 12142481.
- ^ a b c Goodsell DS (1998). The machinery of life. New York: Copernicus. ISBN 978-0-387-98273-1.[페이지 필요]
- ^ a b Paul JR (1971). A History of Poliomyelitis. (Yale studies in the history of science and medicine). New Haven, Conn: Yale University Press. ISBN 978-0-300-01324-5.[페이지 필요]
- ^ "Behind the picture: Rosalind Franklin and the polio model". Medical Research Council. 2019-03-14. Retrieved 4 September 2019.
- ^ Maddox B (2003). Rosalind Franklin: The Dark Lady of DNA. London: Harper Collins. p. 296. ISBN 0-00-655211-0.
- ^ Brown A (2007). J.D. Bernal: The Sage of Science. New York: Oxford University Press. pp. 359–61. ISBN 978-0-19-920565-3.
- ^ Racaniello VR, Baltimore D (August 1981). "Molecular cloning of poliovirus cDNA and determination of the complete nucleotide sequence of the viral genome". Proceedings of the National Academy of Sciences of the United States of America. 78 (8): 4887–91. Bibcode:1981PNAS...78.4887R. doi:10.1073/pnas.78.8.4887. PMC 320284. PMID 6272282.
- ^ Kitamura N, Semler BL, Rothberg PG, Larsen GR, Adler CJ, Dorner AJ, et al. (June 1981). "Primary structure, gene organization and polypeptide expression of poliovirus RNA". Nature. 291 (5816): 547–53. Bibcode:1981Natur.291..547K. doi:10.1038/291547a0. PMID 6264310. S2CID 4352308.
- ^ Hogle JM, Chow M, Filman DJ (27 Sep 1985). "Three-dimensional structure of poliovirus at 2.9 A resolution". Science. 229 (4720): 1358–65. doi:10.1126/science.2994218. PMID 2994218.
- ^ a b c De Jesus NH (July 2007). "Epidemics to eradication: the modern history of poliomyelitis". Virology Journal. 4 (1): 70. doi:10.1186/1743-422X-4-70. PMC 1947962. PMID 17623069.
- ^ Mendelsohn CL, Wimmer E, Racaniello VR (March 1989). "Cellular receptor for poliovirus: molecular cloning, nucleotide sequence, and expression of a new member of the immunoglobulin superfamily". Cell. 56 (5): 855–65. doi:10.1016/0092-8674(89)90690-9. PMID 2538245. S2CID 44296539.
- ^ a b He Y, Mueller S, Chipman PR, Bator CM, Peng X, Bowman VD, et al. (April 2003). "Complexes of poliovirus serotypes with their common cellular receptor, CD155". Journal of Virology. 77 (8): 4827–35. doi:10.1128/JVI.77.8.4827-4835.2003. PMC 152153. PMID 12663789.
- ^ Dunnebacke TH, Levinthal JD, Williams RC (October 1969). "Entry and release of poliovirus as observed by electron microscopy of cultured cells". Journal of Virology. 4 (4): 505–13. doi:10.1128/JVI.4.4.505-513.1969. PMC 375900. PMID 4309884.
- ^ Kaplan G, Freistadt MS, Racaniello VR (October 1990). "Neutralization of poliovirus by cell receptors expressed in insect cells". Journal of Virology. 64 (10): 4697–702. doi:10.1128/JVI.64.10.4697-4702.1990. PMC 247955. PMID 2168959.
- ^ Gómez Yafal A, Kaplan G, Racaniello VR, Hogle JM (November 1993). "Characterization of poliovirus conformational alteration mediated by soluble cell receptors". Virology. 197 (1): 501–05. doi:10.1006/viro.1993.1621. PMID 8212594.
- ^ a b Mueller S, Wimmer E, Cello J (August 2005). "Poliovirus and poliomyelitis: a tale of guts, brains, and an accidental event". Virus Research. 111 (2): 175–93. doi:10.1016/j.virusres.2005.04.008. PMID 15885840.
- ^ Brandenburg B, Lee LY, Lakadamyali M, Rust MJ, Zhuang X, Hogle JM (July 2007). "Imaging poliovirus entry in live cells". PLOS Biology. 5 (7): e183. doi:10.1371/journal.pbio.0050183. PMC 1914398. PMID 17622193.
- ^ Attardi G, Smith J (1962). "Virus specific protein and a ribo-nucleic acid associated with ribosomes in poliovirus infected HeLa cells". Cold Spring Harbor Symposia on Quantitative Biology. 27: 271–92. doi:10.1101/SQB.1962.027.001.026. PMID 13965389.
- ^ Lozano G, Fernandez N, Martinez-Salas E (February 2016). "Modeling Three-Dimensional Structural Motifs of Viral IRES". Journal of Molecular Biology. 428 (5 Pt A): 767–76. doi:10.1016/j.jmb.2016.01.005. PMID 26778619.
- ^ Chen CY, Sarnow P (April 1995). "Initiation of protein synthesis by the eukaryotic translational apparatus on circular RNAs". Science. 268 (5209): 415–17. Bibcode:1995Sci...268..415C. doi:10.1126/science.7536344. PMID 7536344.
- ^ Pelletier J, Sonenberg N (July 1988). "Internal initiation of translation of eukaryotic mRNA directed by a sequence derived from poliovirus RNA". Nature. 334 (6180): 320–25. Bibcode:1988Natur.334..320P. doi:10.1038/334320a0. PMID 2839775. S2CID 4327857.
- ^ Jang SK, Kräusslich HG, Nicklin MJ, Duke GM, Palmenberg AC, Wimmer E (August 1988). "A segment of the 5' nontranslated region of encephalomyocarditis virus RNA directs internal entry of ribosomes during in vitro translation". Journal of Virology. 62 (8): 2636–43. doi:10.1128/JVI.62.8.2636-2643.1988. PMC 253694. PMID 2839690.
- ^ Carter J, Saunders VA (2007). Virology: Principles and Applications. John Wiley & Sons. p. 4. ISBN 978-0-470-02386-0.
- ^ a b Carter J, Saunders VA (2007). Virology: Principles and Applications. John Wiley & Sons. p. 165. ISBN 978-0-470-02386-0.
- ^ Harper DR (2012). Viruses: Biology, Applications, and Control. The United States of America: Garland Science. ISBN 978-0-8153-4150-5.[페이지 필요]
- ^ "Poliomyelitis". Brown University. Archived from the original on February 22, 2007.
- ^ Carter J, Saunders VA (2007). Virology: Principles and Applications. John Wiley & Sons. p. 164. ISBN 978-0-470-02386-0.
- ^ Louten J (2016). "Poliovirus". Essential Human Virology. Elsevier. pp. 257–71. doi:10.1016/b978-0-12-800947-5.00014-4. ISBN 978-0-12-800947-5.
- ^ Murray KE, Barton DJ (April 2003). "Poliovirus CRE-dependent VPg uridylylation is required for positive-strand RNA synthesis but not for negative-strand RNA synthesis". Journal of Virology. 77 (8): 4739–50. doi:10.1128/JVI.77.8.4739-4750.2003. PMC 152113. PMID 12663781.
- ^ Goodfellow IG, Kerrigan D, Evans DJ (January 2003). "Structure and function analysis of the poliovirus cis-acting replication element (CRE)". RNA. 9 (1): 124–37. doi:10.1261/rna.2950603. PMC 1370376. PMID 12554882.
- ^ Carter J, Saunders VA (2007). Virology: Principles and Applications. John Wiley & Sons. pp. 161, 165. ISBN 978-0-470-02386-0.
- ^ Carter J, Saunders VA (2007). Virology: Principles and Applications. John Wiley & Sons. p. 166. ISBN 978-0-470-02386-0.
- ^ a b c d Kew OM, Sutter RW, de Gourville EM, Dowdle WR, Pallansch MA (2005). "Vaccine-derived polioviruses and the endgame strategy for global polio eradication". Annual Review of Microbiology. 59: 587–635. doi:10.1146/annurev.micro.58.030603.123625. PMID 16153180.
- ^ Drake JW (August 1958). "Interference and multiplicity reactivation in polioviruses". Virology. 6 (1): 244–64. doi:10.1016/0042-6822(58)90073-4. PMID 13581529.
- ^ Kirkegaard K, Baltimore D (November 1986). "The mechanism of RNA recombination in poliovirus". Cell. 47 (3): 433–43. doi:10.1016/0092-8674(86)90600-8. PMC 7133339. PMID 3021340.
- ^ Barr JN, Fearns R (June 2010). "How RNA viruses maintain their genome integrity". The Journal of General Virology. 91 (Pt 6): 1373–87. doi:10.1099/vir.0.020818-0. PMID 20335491.
- ^ Bernstein H, Bernstein C, Michod RE (January 2018). "Sex in microbial pathogens". Infection, Genetics and Evolution. 57: 8–25. doi:10.1016/j.meegid.2017.10.024. PMID 29111273.
- ^ Jiang P, Faase JA, Toyoda H, Paul A, Wimmer E, Gorbalenya AE (May 2007). "Evidence for emergence of diverse polioviruses from C-cluster coxsackie A viruses and implications for global poliovirus eradication". Proceedings of the National Academy of Sciences of the United States of America. 104 (22): 9457–62. Bibcode:2007PNAS..104.9457J. doi:10.1073/pnas.0700451104. PMC 1874223. PMID 17517601.
- ^ Jorba J, Campagnoli R, De L, Kew O (May 2008). "Calibration of multiple poliovirus molecular clocks covering an extended evolutionary range". Journal of Virology. 82 (9): 4429–40. doi:10.1128/JVI.02354-07. PMC 2293050. PMID 18287242.
- ^ Rothberg PG, Wimmer E (December 1981). "Mononucleotide and dinucleotide frequencies, and codon usage in poliovirion RNA". Nucleic Acids Research. 9 (23): 6221–29. doi:10.1093/nar/9.23.6221. PMC 327599. PMID 6275352.
- ^ Zhang J, Wang M, Liu WQ, Zhou JH, Chen HT, Ma LN, et al. (March 2011). "Analysis of codon usage and nucleotide composition bias in polioviruses". Virology Journal. 8: 146. doi:10.1186/1743-422X-8-146. PMC 3079669. PMID 21450075.
- ^ "Global eradication of wild poliovirus type 2 declared". Global Polio Eradication Initiative. 2015-09-20. Retrieved 2015-09-30.
- ^ "GPEI-Two out of three wild poliovirus strains eradicated". Retrieved 2019-11-02.
- ^ Carstens EB, Ball LA (July 2009). "Ratification vote on taxonomic proposals to the International Committee on Taxonomy of Viruses (2008)". Archives of Virology. 154 (7): 1181–88. doi:10.1007/s00705-009-0400-2. PMC 7086627. PMID 19495937.
- ^ Mueller S, Wimmer E (August 2003). "Recruitment of nectin-3 to cell-cell junctions through trans-heterophilic interaction with CD155, a vitronectin and poliovirus receptor that localizes to alpha(v)beta3 integrin-containing membrane microdomains". The Journal of Biological Chemistry. 278 (33): 31251–60. doi:10.1074/jbc.M304166200. PMID 12759359.
- ^ Suzuki Y (May 2006). "Ancient positive selection on CD155 as a possible cause for susceptibility to poliovirus infection in simians". Gene. 373: 16–22. doi:10.1016/j.gene.2005.12.016. PMID 16500041.
- ^ Bodian D, Horstmann DH (1969). Polioviruse. Philadelphia, Penn: Lippincott. pp. 430–73.
- ^ Sabin AB (June 1956). "Pathogenesis of poliomyelitis; reappraisal in the light of new data". Science. 123 (3209): 1151–57. Bibcode:1956Sci...123.1151S. doi:10.1126/science.123.3209.1151. PMID 13337331.
- ^ Carter JB, Saunders VA (2007). Virology: Principles and Applications (1st ed.). Chichester, England: John Wiley & Sons. ISBN 978-0-470-02386-0. OCLC 124160564.[페이지 필요]
- ^ e메디컬의 급성 폴리오밀염
eMedical의 소아과 폴리오밀염 - ^ Yang WX, Terasaki T, Shiroki K, Ohka S, Aoki J, Tanabe S, et al. (March 1997). "Efficient delivery of circulating poliovirus to the central nervous system independently of poliovirus receptor". Virology. 229 (2): 421–28. doi:10.1006/viro.1997.8450. PMID 9126254.
- ^ Ohka S, Yang WX, Terada E, Iwasaki K, Nomoto A (October 1998). "Retrograde transport of intact poliovirus through the axon via the fast transport system". Virology. 250 (1): 67–75. doi:10.1006/viro.1998.9360. PMID 9770421.
- ^ Ren R, Racaniello VR (October 1992). "Poliovirus spreads from muscle to the central nervous system by neural pathways". The Journal of Infectious Diseases. 166 (4): 747–52. doi:10.1093/infdis/166.4.747. PMID 1326581.
- ^ Lancaster KZ, Pfeiffer JK (March 2010). Gale Jr M (ed.). "Limited trafficking of a neurotropic virus through inefficient retrograde axonal transport and the type I interferon response". PLOS Pathogens. 6 (3): e1000791. doi:10.1371/journal.ppat.1000791. PMC 2832671. PMID 20221252.
- ^ Ida-Hosonuma M, Iwasaki T, Yoshikawa T, Nagata N, Sato Y, Sata T, et al. (April 2005). "The alpha/beta interferon response controls tissue tropism and pathogenicity of poliovirus". Journal of Virology. 79 (7): 4460–69. doi:10.1128/JVI.79.7.4460-4469.2005. PMC 1061561. PMID 15767446.
- ^ Ohka S, Igarashi H, Nagata N, Sakai M, Koike S, Nochi T, et al. (August 2007). "Establishment of a poliovirus oral infection system in human poliovirus receptor-expressing transgenic mice that are deficient in alpha/beta interferon receptor". Journal of Virology. 81 (15): 7902–12. doi:10.1128/JVI.02675-06. PMC 1951287. PMID 17507470.
- ^ Racaniello VR (January 2006). "One hundred years of poliovirus pathogenesis". Virology. 344 (1): 9–16. doi:10.1016/j.virol.2005.09.015. PMID 16364730.
- ^ Carter JB, Saunders VA (2007). Virology: Principles and Applications. Liverpool John Moores University, UK: John Wiley & Sons. ISBN 978-0-470-02386-0.[페이지 필요]
- ^ "WHO Poliomyelitis". Archived from the original on May 17, 2014.
- ^ Ren RB, Costantini F, Gorgacz EJ, Lee JJ, Racaniello VR (October 1990). "Transgenic mice expressing a human poliovirus receptor: a new model for poliomyelitis". Cell. 63 (2): 353–62. doi:10.1016/0092-8674(90)90168-E. PMID 2170026. S2CID 11946113.
- ^ Koike S, Taya C, Kurata T, Abe S, Ise I, Yonekawa H, Nomoto A (February 1991). "Transgenic mice susceptible to poliovirus". Proceedings of the National Academy of Sciences of the United States of America. 88 (3): 951–55. Bibcode:1991PNAS...88..951K. doi:10.1073/pnas.88.3.951. PMC 50932. PMID 1846972.
- ^ Horie H, Koike S, Kurata T, Sato-Yoshida Y, Ise I, Ota Y, et al. (February 1994). "Transgenic mice carrying the human poliovirus receptor: new animal models for study of poliovirus neurovirulence". Journal of Virology. 68 (2): 681–88. doi:10.1128/JVI.68.2.681-688.1994. PMC 236503. PMID 8289371.
- ^ Ohka S, Nomoto A (October 2001). "Recent insights into poliovirus pathogenesis". Trends in Microbiology. 9 (10): 501–06. doi:10.1016/S0966-842X(01)02200-4. PMID 11597452.
- ^ Koike S, Taya C, Aoki J, Matsuda Y, Ise I, Takeda H, et al. (1994). "Characterization of three different transgenic mouse lines that carry human poliovirus receptor gene--influence of the transgene expression on pathogenesis". Archives of Virology. 139 (3–4): 351–63. doi:10.1007/BF01310797. PMID 7832641. S2CID 11070024.
- ^ a b Nagata N, Iwasaki T, Ami Y, Sato Y, Hatano I, Harashima A, et al. (March 2004). "A poliomyelitis model through mucosal infection in transgenic mice bearing human poliovirus receptor, TgPVR21". Virology. 321 (1): 87–100. doi:10.1016/j.virol.2003.12.008. PMID 15033568.
- ^ Dragunsky E, Nomura T, Karpinski K, Furesz J, Wood DJ, Pervikov Y, et al. (2003). "Transgenic mice as an alternative to monkeys for neurovirulence testing of live oral poliovirus vaccine: validation by a WHO collaborative study". Bulletin of the World Health Organization. 81 (4): 251–60. PMC 2572431. PMID 12764491.
- ^ Racaniello VR, Baltimore D (November 1981). "Cloned poliovirus complementary DNA is infectious in mammalian cells". Science. 214 (4523): 916–19. Bibcode:1981Sci...214..916R. doi:10.1126/science.6272391. PMID 6272391.
- ^ Cello J, Paul AV, Wimmer E (August 2002). "Chemical synthesis of poliovirus cDNA: generation of infectious virus in the absence of natural template". Science. 297 (5583): 1016–18. Bibcode:2002Sci...297.1016C. doi:10.1126/science.1072266. PMID 12114528. S2CID 5810309.
- ^ Couzin J (July 2002). "Virology. Active poliovirus baked from scratch". Science. 297 (5579): 174–75. doi:10.1126/science.297.5579.174b. PMID 12114601. S2CID 83531627.
- ^ Brown MC, Dobrikova EY, Dobrikov MI, Walton RW, Gemberling SL, Nair SK, et al. (November 2014). "Oncolytic polio virotherapy of cancer". Cancer. 120 (21): 3277–86. doi:10.1002/cncr.28862. PMC 4205207. PMID 24939611.
외부 링크
- ICTVdb 바이러스 분류 2006
- 피코르나비루스의 본고장(종, 세로타입 및 제안된 변경사항의 최신 업데이트) 2010-11-04년 Wayback Machine에 보관
- Goodsell D. "Poliovirus and Rhinovirus". August 2001 Molecule of the Month.
- 전자파 데이터 뱅크(EMDB)에 보관된 소아마비 바이러스의 3D 고분자 구조
- "Human poliovirus 1". NCBI Taxonomy Browser. 12080.
- "Human poliovirus 3". NCBI Taxonomy Browser. 12086.




