인간 진화 유전학
Human evolutionary genetics인간 진화 유전학은 한 인간의 게놈이 다른 인간 게놈과 어떻게 다른지, 인간 게놈을 발생시킨 진화적 과거, 그리고 그 현재의 영향을 연구한다. 게놈들 간의 차이는 인류학, 의학, 역사적, 법의학적 함의와 응용을 가지고 있다. 유전자 데이터는 인간 진화에 대한 중요한 통찰력을 제공할 수 있다.
유인원의 기원
−10 — – −9.5 — – −9 — – −8.5 — – −8 — – −7.5 — – −7 — – −6.5 — – −6 — – −5.5 — – −5 — – −4.5 — – −4 — – −3.5 — – −3 — – −2.5 — – −2 — – −1.5 — – −1 — – −0.5 — – 0 — |
| |||||||||||||||||||
(백만년 전) | ||||||||||||||||||||
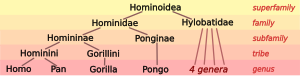
생물학자들은 인간을 몇몇 다른 종들과 함께 거대한 유인원(호미니과 가족의 종)으로 분류한다. 살아 있는 호미니과는 뚜렷한 두 종의 침팬지(보노보, 파니쿠스, 침팬지, 판트로글로디테스), 두 종의 고릴라(서구 고릴라, 동부 고릴라, 고릴라 그라워리), 두 종의 오랑우탄(보르네안 오랑우탄, 퐁고 아벨리)이 있다. 기븐족 히로바티과와 함께 있는 유인원은 유인원의 슈퍼 호미노이데아를 형성한다.
유인원은 차례로 구세계원숭이, 신대륙원숭이 등과 함께 영장류(>400종)에 속한다. 미토콘드리아 DNA(mtDNA)와 핵 DNA(nDNA) 양쪽의 데이터는 영장류가 로덴티아, 라고모르파, 더모프테라, 스칸덴티아와 함께 에우아콘톨리우스 그룹에 속한다는 것을 나타낸다.[1] 이는 에우아르콘토글리어의 회원들에서만 발견된 알루처럼 짧은 변위 핵원소(SINEs)에 의해 더욱 뒷받침된다.[2]
계통유전학
계통생식 나무는 대개 개체군으로부터 DNA나 단백질 서열에서 파생된다. 종종 미토콘드리아 DNA나 Y염색체 염기서열은 고대 인간의 인구통계학을 연구하는 데 사용된다. DNA의 이러한 단일 로쿠스 출처는 재결합하지 않으며 mtDNA에서 단 하나의 예외만을 가지고 거의 항상 단일 부모로부터 유전된다.[3] 더 가까운 지리적 지역의 개인은 일반적으로 더 먼 지역의 개개인보다 더 유사한 경향이 있다. 계통생성 트리의 거리는 다음을 나타내기 위해 대략 사용할 수 있다.
- 유전적 거리. 인간과 침팬지의 유전적 차이는 2% [4]미만으로 현대인간의 변화(0.6%로 추정)의 3배에 이른다.[5]
- 가장 최근의 공통 조상의 일시적 고립성. 현대 인류의 가장 최근의 공통 조상은 대략 16만년 전에 살았던 것으로 추정되며,[6] 인간과 침팬지의 최근 공통 조상은 대략 5백만년에서 6백만년 전에 살았던 것으로 추정된다.[7]
인간과 아프리카 유인원의 분류
인간과 가장 가까운 친척인 비인간 유인원(침팬지와 고릴라)과의 분리는 1세기 이상 광범위하게 연구되어 왔다. 다음과 같은 5가지 주요 질문이 제시되었다.
- 우리의 가장 가까운 조상은 어떤 유인원인가?
- 언제 분리가 일어났는가?
- 분할 전 공통 조상의 유효 모집단 크기는 얼마였습니까?
- 분화 전 인구구조(하위) 또는 부분혼화물이 그 뒤를 잇고 있는 흔적이 있는가?
- 분리 전과 그 후의 구체적인 사건(염색체 2a와 2b의 융합 포함)은 무엇이었는가?
일반 관측치
앞서 논의한 바와 같이 게놈의 다른 부분은 서로 다른 호미노이드 사이의 다른 시퀀스 차이를 보여준다. 인간과 침팬지의 DNA의 염기서열 차이도 큰 것으로 나타났다. 예를 들어 인간과 침팬지의 비코딩, 비반복성 유전체 영역 간에 시퀀스 차이가 0%에서 2.66%까지 다양하다.[8] 침팬지 게놈(판트로6)에서 일대일 정확히 일치하는 인간 게놈(hg38)의 뉴클레오티드 비율은 84.38%로 나타났다. 또한 DNA 세그먼트의 비교 분석에 의해 생성된 유전자 트리가 종 트리에 항상 맞는 것은 아니다. 요약:
- 그 순서의 차이는 인간, 침팬지, 고릴라 사이에 현저하게 다르다.
- 대부분의 DNA 서열에서 인간과 침팬지는 가장 밀접하게 연관되어 있는 것으로 보이지만, 어떤 이들은 인간과 고릴라 또는 침팬지-고릴라 성질을 가리킨다.
- 침팬지 게놈뿐만 아니라 인간의 게놈도 서열화되었다. 인간은 23쌍의 염색체를 가지고 있는 반면 침팬지, 고릴라, 오랑우탄은 24쌍을 가지고 있다. 인간 염색체 2는 다른 영장류에서 분리되어 남아 있던 2a와 2b의 두 염색체가 융합된 것이다.[9]
발산 시간
인간이 다른 유인원으로부터 발산하는 시간은 매우 흥미롭다. 1967년에 발표된 최초의 분자 연구 중 하나는 다른 영장류 사이의 면역학적 거리(IDs)를 측정했다.[10] 기본적으로 이 연구는 한 종(인간 알부민)의 항원이 다른 종(인간, 침팬지, 고릴라, 구세계 원숭이)의 면역 체계에서 유도하는 면역학적 반응의 강도를 측정했다. 밀접하게 연관된 종들은 유사한 항원을 가지고 있어야 하고 따라서 서로의 항원에 대한 면역학적 반응이 약해야 한다. 자신의 항원(예: 인간 대 인간)에 대한 종의 면역학적 반응은 1로 설정되었다.
인간과 고릴라 사이의 ID는 1.09로, 인간과 침팬지 사이의 ID는 1.14로 결정되었다. 그러나 6마리의 다른 구세계 원숭이와의 거리는 평균 2.46으로 아프리카 유인원이 원숭이보다 인간과 더 밀접한 관계가 있음을 보여준다. 저자들은 화석 자료를 토대로 구세계원숭이와 호미노이드의 발산 시간을 3000만년 전(MYA)으로 보고 면역학적 거리는 일정한 비율로 성장한다고 봤다. 그들은 인간과 아프리카 유인원의 발산 시간이 대략 5 MYA 정도라고 결론지었다. 그것은 놀라운 결과였다. 당시 대부분의 과학자들은 인간과 유인원이 훨씬 일찍 분리되었다고 생각했다(>15 MYA).
고릴라는 신분증상으로 침팬지보다 인간과 더 가까웠지만, 그 차이가 너무 미미해 삼초절개술을 확실하게 해결할 수 없었다. 나중에 분자유전학에 기초한 연구는 삼분자유전학을 해결할 수 있었다: 침팬지는 고릴라보다 유전학적으로 인간과 더 가깝다. 그러나, (분자 유전학에서 훨씬 더 정교한 방법을 사용하여) 나중에 추정된 일부 발산 시간은 1967년 최초의 추정치와 실질적으로 다르지 않지만, 최근의 한 논문에서는[11] 이를 11–14 MYA로 제시하였다.
다양성 시간 및 조상 유효 인구 크기
발산 시간을 결정하는 현재의 방법은 DNA 시퀀스 정렬과 분자 시계를 사용한다. 보통 분자시계는 아프리카 유인원(인간 포함) 12-16 MYA에서 갈라진 오랑우탄을 가정하여 보정된다. 일부 연구에는 일부 구세계 원숭이가 포함되어 있으며, 호미노이드에서 25-30 MYA로 분자시계를 설정하기도 한다. 두 교정점 모두 극히 적은 화석 자료를 바탕으로 하고 있으며 비난을 받아왔다.[12]
이 날짜들이 수정되면 분자 데이터에서 추정된 발산 시간 또한 바뀔 것이다. 그러나 상대적 발산 시간은 달라질 것 같지 않다. 절대 발산 시간을 정확히 알 수는 없더라도 침팬지와 사람 사이의 발산 시간은 침팬지(또는 인간)와 원숭이 사이의 차이보다 약 6배 짧다는 것을 꽤 확신할 수 있다.
한 연구(타카하타 외, 1995년)는 인간과 침팬지의 게놈의 서로 다른 영역에서 15개의 DNA 시퀀스와 인간, 침팬지, 고릴라의 7개의 DNA 시퀀스를 사용했다.[13] 그들은 침팬지가 고릴라보다 인간과 더 밀접하게 연관되어 있다고 판단했다. 다양한 통계적 방법을 사용하여, 그들은 인간-침팬지의 발산 시간을 4.7 MYA로, 고릴라와 인간(그리고 침팬지) 사이의 발산 시간을 7.2 MYA로 추정했다.
또한 그들은 인간과 침팬지의 공통 조상의 유효 개체 수를 10만 마리로 추정했다. 이것은 오늘날 인간의 유효한 인구 규모가 1만 명에 불과할 것으로 추정되기 때문에 다소 놀라운 일이었다. 만약 그것이 사실이라면, 인간의 혈통이 그것의 진화에서 그것의 유효 인구 크기(따라서 유전적 다양성)의 엄청난 감소를 경험했을 것이라는 것을 의미한다. (토바 재앙론 참조)

또 다른 연구(Chen & Li, 2001년)는 인간, 침팬지, 고릴라, 오랑우탄에서 53개의 비반복적이고 유전적인 DNA를 추출했다.[8] DNA 서열을 긴 한 시퀀스에 연결했을 때, 생성된 이웃접착 나무는 100% 부스트랩으로 호모판 쇄도를 지지했다(즉 인간과 침팬지는 네 종 중에서 가장 가까운 관련 종이다). 세 종(인간, 침팬지, 고릴라 등)이 서로 상당히 밀접하게 연관되어 있을 때, DNA 시퀀스 데이터로부터 얻은 나무는 특정화(종목)를 나타내는 나무와 합치되지 않을 수 있다.
내부 시간 범위(TIN)가 짧을수록 부조화 유전자 트리가 흔하다. 내부 모집단의 유효 모집단 크기(Ne)는 모집단에서 유전적 선형이 얼마나 오래 보존되는지를 결정한다. 더 높은 유효 인구 크기는 더 많은 조화되지 않은 유전자 나무를 야기한다. 따라서 만약 내부 시간범위가 알려지면 인간과 침팬지의 공통 조상의 조상의 유효인구 규모를 계산할 수 있다.
각 세그먼트를 개별적으로 분석했을 때 31개가 호모판 클래드를 지지했고, 10개는 호모-고릴라 클래드를 지지했으며, 12개는 판고릴라 클래드를 지지했다. 저자들은 분자 시계를 이용해 고릴라가 처음 6.2-8.4 MYA로 갈라졌고 침팬지와 인간은 160-220만년 후(내부 시간 범위) 4.6-6.2 MYA로 갈라졌다고 추정했다. 내부 시간 범위는 인간과 침팬지의 공통 조상의 효과적인 인구 규모를 추정하는 데 유용하다.
단순 분석 결과 24 loci가 호모 판의 쇄도를 지지했고 7명이 호모-고릴라 쇄도를 지지했으며 2명은 범고릴라 쇄도를 지지했으며 20명은 아무런 결의도 하지 않았다. 또한 그들은 데이터베이스에서 35개의 단백질 코딩 로키를 가져갔다. 이 12개 중 호모판 쇄도를 지지한 12개, 호모-고릴라 쇄도 3개, 범고릴라 쇄도 4개, 그리고 16개는 아무런 결의도 하지 않았다. 따라서 해상도를 부여한 52개 loci 중 ~70%(유전자간 33개, 단백질 부호화 19개)만이 '올바른' 종목을 지원한다. 종나무를 지원하지 않는 loci의 분수와 이전에 그들이 추정했던 내부 시간 범위로부터 인간과 침팬지의 공통 조상의 유효인구는 약 52,000 - 96,000명으로 추정되었다. 이 값은 제1차 연구(타카하타)의 값만큼 높은 것이 아니라, 여전히 오늘날 인간의 유효인구 규모보다 훨씬 높은 것이다.
세 번째 연구(양, 2002년)는 천과 리가 사용했던 것과 동일한 데이터 집합을 사용했지만 다른 통계적 방법을 사용하여 조상 유효인구를 '1만2000명' ~2만1000명으로 추정했다.[14]
인간과 다른 유인원 사이의 유전적 차이
인간과 침팬지의 게놈 안에서 정렬 가능한 염기서열은 단일 뉴클레오티드 대체물 약 3,500만 개씩 차이가 난다. 또한 전체 게놈의 약 3%는 삭제, 삽입, 중복에 의해 다르다.[15]
변이율은 비교적 일정하기 때문에, 이러한 변화의 약 절반은 인간 혈통에 일어났다. 이러한 고정된 차이점들 중 극히 일부만이 인간과 침팬지의 다른 표현형태를 만들어 냈고 그것들을 발견하는 것은 큰 도전이다. 대부분의 차이점들은 중립적이며 표현형에는 영향을 미치지 않는다.[citation needed]
분자 진화는 단백질 진화, 유전자 손실, 미분 유전자 조절, RNA 진화를 통해 다른 방식으로 작용할 수 있다. 모두 인간 진화에 어느 정도 작용한 것으로 생각된다.
유전자손실
많은 다른 돌연변이는 유전자를 비활성화할 수 있지만, 특정한 방식으로 그 기능을 변화시킬 수 있는 것은 거의 없다. 따라서 비활성 돌연변이는 쉽게 선택할 수 있게 될 것이다. 따라서 유전자 손실은 진화 적응의 일반적인 메커니즘이 될 수 있다("더 적은" 가설").[16]
인간의 혈통에서 80개의 유전자가 침팬지와 마지막 공통의 조상으로부터 분리되어 없어졌다. 그 중 36개는 후각 수용체용이었다. 화학적 감각과 면역 반응에 관여하는 유전자는 과잉 대표된다.[17] 또 다른 연구는 86개의 유전자가 손실된 것으로 추정했다.[18]
헤어 케라틴 유전자 KRTHAP1
1형 머리 케라틴의 유전자는 인간의 혈통에서 사라졌다. 케라틴은 머리카락의 주요 성분이다. 인간은 여전히 9가지 기능적 I형 머리 케라틴 유전자를 가지고 있지만, 그 특정 유전자의 상실이 인간의 체모 털을 얇게 만드는 원인이 되었을지도 모른다. 이 연구는 일정한 분자 시계를 가정해 볼 때, 인류 진화에 있어서 비교적 최근에 발생한 유전자 손실은 - 240,000년도 채 되지 않았지만 - 빈디자 네안데르탈과 높은 탐지거리 데니소반 순서는 모두 현대 인간과 같은 조급 정지 코돈들을 포함하고 있으므로, 데이트는 a 750,000년 이상이어야 한다.가거라
미오신유전자MYH16
스테드먼 외 (2004)는 인간의 혈통에서 성체 미오신 유전자 MYH16의 상실로 인해 수축근육이 작아졌다고 밝혔다. 그들은 아프리카에서 호모 에가스터/에레토스의 출현을 앞지르면서 불활성화(두 개의 염기쌍 삭제)로 이어진 돌연변이가 240만년 전에 일어났다고 추정했다. 그 뒤의 기간은 두개골 용량의 강한 증가가 특징으로, 유전자의 손실로 인해 호모 속에서의 뇌 크기에 대한 진화적 제약이 제거되었을 것이라는 추측을 불러일으켰다.[20]
MYH16 유전자의 손실에 대한 또 다른 추정치는 호모가 나타나기 훨씬 전인 530만년 전이다.[21]
기타
유전자 덧셈
부분복제(SD 또는 LCR)는 새로운 영장류 유전자를 만들고 인간의 유전적 변이를 형성하는 역할을 해왔다.
사람별 DNA 삽입
인간 게놈을 침팬지, 고릴라, 오랑우탄, 긴팔원숭이, 마카크 등 비교 영장류 5종의 게놈과 비교했을 때, 규제 대상으로 추정되는 인간 고유의 삽입물이 약 2만 개에 이르는 것으로 나타났다. 대부분의 삽입물이 피트니스 중립인 것처럼 보이지만, 신경 표현형 및 치과 및 감각 지각과 관련된 표현형과 관련된 몇몇 연관성을 보여주는 선택된 유전자에서 소량이 확인되었다. 이러한 발견들은 최근 인간의 진화에서 인간 특유의 삽입이 중요하게 보이는 역할을 암시한다.[22]
선택 압력
인간의 가속 영역은 인간과 침팬지가 공통의 조상을 가지고 있었기 때문에 그 시간 동안 유전적 표류로 설명될 수 있는 것보다 훨씬 더 큰 정도로 게놈의 영역이다. 이들 지역은 자연선택의 대상이 되어 뚜렷이 인간적 특성의 진화를 이끌어 낼 조짐을 보이고 있다. 뇌 발달과 관련이 있다고 여겨지는 HAR1F와 반대되는 엄지손가락의 발달에 역할을 했을지도 모르는 HAR2(즉, HACNS1)가 두 가지 예다.
인간과 침팬지의 차이가 상당 부분 유전자 자체의 차이보다 유전자 발현 조절에 기인한다는 가설도 나왔다. 종종 기능적이고 따라서 긍정적으로 선택된 규제 영역을 포함하는 보존된 비코딩 시퀀스의 분석은 이러한 가능성을 다룬다.[23]
인간과 유인원 사이의 염기서열 차이
2005년 여름 일반 침팬지(Pan troglodytes) 게놈의 초안 순서가 발표되었을 때 인간 게놈과 비교할 수 있을 정도로 2억 4천만 염기(약 3억 1천만 염기)의 염기서열이 잘 조립되었고, 이 염기서열의 1.23%는 단일 염기 대체에 의해 차이가 났다.[15] 이 중 1.06% 이하는 종간의 고정된 차이를 나타낸다고 생각되었고, 나머지는 인간이나 침팬지의 변형된 장소였다. 인델(삽입/삭제)이라고 불리는 또 다른 형태의 차이는 많은 차이를 덜 차지하지만(15% 만큼 많음) 각각의 게놈에 고유한 순서의 1.5%를 기여했다. 각 게놈은 한 베이스에서 수백만 베이스까지 모든 베이스를 포함할 수 있기 때문이다.[15]
한 동반 논문에서 두 게놈의 부분복제를 조사했는데,[24] 게놈에 삽입하고 삭제하는 것이 지워지지 않는 염기서열의 대부분을 차지한다. 그들은 유색 계열의 총 2.7%가 한 혈통 또는 다른 혈통에서 서로 다르게 복제되었다는 것을 발견했다.
| 로커스 | 휴먼-침프 | 휴먼고릴라 | 휴먼오랑우탄 |
|---|---|---|---|
| 알루 원소 | 2 | - | - |
| 비코딩(Chr. Y) | 1.68 ± 0.19 | 2.33 ± 0.2 | 5.63 ± 0.35 |
| 유사생성(자동) | 1.64 ± 0.10 | 1.87 ± 0.11 | - |
| 유사생식(Chr. X) | 1.47 ± 0.17 | - | - |
| 비코딩(자동) | 1.24 ± 0.07 | 1.62 ± 0.08 | 3.08 ± 0.11 |
| 유전자(Ks) | 1.11 | 1.48 | 2.98 |
| 인트론스 | 0.93 ± 0.08 | 1.23 ± 0.09 | - |
| Xq13.3 | 0.92 ± 0.10 | 1.42 ± 0.12 | 3.00 ± 0.18 |
| X염색체 소계 | 1.16 ± 0.07 | 1.47 ± 0.08 | - |
| 유전자(Ka) | 0.8 | 0.93 | 1.96 |
시퀀스 차이에는 일반적으로 다음과 같은 패턴이 있다. 휴먼-침프 < 휴먼-고릴라> <휴먼-오랑우탄>은 인간과 아프리카 유인원 사이의 긴밀한 친분을 부각시켰다. 알루 원소는 게놈의 평균 뉴클레오티드보다 약 10배 더 자주 돌연변이를 일으키는 CpG 디뉴클레오티드의 높은 빈도 때문에 빠르게 분화한다. 변이율은 남성 생식선에서 더 높기 때문에 아버지에게서만 유전되는 Y염색체의 분비가 오토솜에서보다 더 높다. X염색체는 남성 생식선보다 여성 생식선을 통해 2배 더 자주 유전되기 때문에 염기서열이 약간 낮은 분열을 보인다. Xq13.3 지역의 시퀀스 차이는 인간과 침팬지 사이에서 놀라울 정도로 낮다.[25]
단백질(Ka)의 아미노산 염기서열을 바꾸는 돌연변이는 가장 흔하지 않다. 사실 모든 직교 단백질의 29%는 인간과 침팬지 사이에서 동일하다. 전형적인 단백질은 단지 두 개의 아미노산에 의해 다르다.[15] 표에 나타난 시퀀스 차이 측정은 예를 들어 A(아데닌)에서 G(게아닌)까지의 대체 차이만 고려한다. 그러나 DNA 시퀀스는 베이스 삽입 및 삭제(인델)에 따라 다를 수 있다. 이것들은 일반적으로 시퀀스 차이 계산이 수행되기 전에 선형에서 벗겨진다.
현대 인간과 네안데르탈인의 유전적 차이
국제 과학자 그룹은 2010년 5월에 네안데르탈인 게놈의 초안을 완성했다. 아프리카인이 아닌 사람들의 게놈은 아프리카계 아프리카인의 게놈보다 네안데르탈인과 1-4% 더 공통점이 있기 때문에, 그 결과는 현대인(호모 사피엔스)과 네안데르탈인(호모 네안데르탈렌시스) 사이의 일부 번식을 나타낸다. 네안데르탈인과 대부분의 현대인들은 젖을 뗀 후 젖당을 분해할 수 없는 효소를 인코딩하는 락타아제 유전자의 유당 내성 변종을 공유한다. 현대 인간과 네안데르탈인은 또한 뇌 발달과 현대 인간의 언어와 관련된 FOXP2 유전자 변종을 공유하는데, 이는 네안데르탈인이 말을 할 수 있었음을 보여준다. 침팬지는 인간과 네안데르탈인 FOXP2에 비해 FOXP2에서 아미노산 차이가 두 가지 있다.[26][27][28]
현대인간의 유전적 차이
H. 사피엔스는 약 30만년 전에 출현한 것으로 생각된다. 그것은 아프리카 전역으로, 그리고 7만년 전 유라시아와 오세아니아 전역으로 흩어졌다. 2009년 한 연구는 14개의 "동향 인구 집단"을 확인했는데, 가장 멀리 떨어진 곳은 남아프리카 공화국의 산족이었다.[29][30]
다양한 기후 지역에 걸쳐 급속도로 팽창하고, 특히 소의 가축화와 농업의 발달로 인해, 인류는 분산된 이후 상당한 선택적 압력에 노출되어 왔다. 예를 들어, EDAR, ADH1B, ABCC1, ALDH2genes의 변형을 포함한 선택 압력을 암시하는 다수의 농축된 알레르기에 의해 동아시아인이 유로피드와 분리되어 있는 것으로 밝혀졌다. 특히 동아시아 타입의 ADH1B는 쌀 재배와 연관되어 있으며, 따라서 약 1만년 전에 쌀 재배가 발달한 후에 발생하였을 것이다.[31] 동아시아인의 특징에 대한 여러 표현적 특성들은 3만 5천년 전의 EDAR 유전자의 단일 돌연변이 때문이다.[32]
2017년[update] 현재 SNP 등 변형을 나열한 단일 뉴클레오티드 다형성 데이터베이스(dbSNP)에는 서열화된 인간 게놈에서 발견된 총 3억2400만 개의 변종이 나열돼 있다.[33] 두 개인 간에 차이가 나는 뉴클레오티드의 평균 비율인 뉴클레오티드의 다양성은 동시대의 인간(인간과 침팬지 사이의 2%에 비해)이 0.1%에서 0.4%로 추정된다.[34][35] 이는 수백만 개의 사이트에서 게놈의 차이와 일치한다. 1000개의 게놈 프로젝트도 마찬가지로 "일반적인 [개별] 게놈은 410만 개에서 550만 개로 참조 인간 게놈과 다르며, 이는 2천만 개의 염기서열에도 영향을 미친다."[36]라고 밝혔다.
2019년 2월 과학자들은 인공지능(AI)을 이용한 유전학 연구를 바탕으로 현대인간의 게놈에서 네안데르탈인, 데니소반, 인간혼합물(하이브리드 호민)이 아닌 미지의 인류 조상종의 존재를 암시하는 증거를 발견했다.[37][38]
연구 연구
연구진에 따르면 2019년 3월 중국 과학자들은 인간의 뇌와 관련된 MCPH1 유전자를 실험용 붉은털원숭이에 삽입해 유전자변형원숭이가 비전형원숭이를 제어하는 것에 비해 '색상과 모양이 일치하는 단기 기억력 테스트'에서 더 좋은 성과를 내고 더 빠른 답을 얻었다고 보고했다.[39][40]
참고 항목
참조
- ^ Murphy, W.J.; Eizirik, E.; O'Brien, S.J.; Madsen, O.; Scally, M.; Douady, C.J.; Teeling, E.; Ryder, O.A.; Stanhope, M.J.; de Jong, W.W.; Springer, M.S. (2001). "Resolution of the early placental mammal radiation using Bayesian phylogenetics". Science. 294 (5550): 2348–2351. Bibcode:2001Sci...294.2348M. doi:10.1126/science.1067179. PMID 11743200. S2CID 34367609.
- ^ Kriegs, J.O.; Churakov, G.; Kiefmann, M.; Jordan, U.; Brosius, J.; Schmitz, J. (2006). "Retroposed elements as archives for the evolutionary history of placental mammals". PLOS Biol. 4 (4): e91. doi:10.1371/journal.pbio.0040091. PMC 1395351. PMID 16515367.
- ^ Schwartz M, Vissing J (2002). "Paternal Inheritance of Mitochondrial DNA". N Engl J Med. 347 (8): 576–580. doi:10.1056/NEJMoa020350. PMID 12192017.
- ^ "인간 염색체 2." PBS
- ^ 2015년 기준, 두 개인의 게놈의 일반적인 차이는 2,000만 염기쌍(또는 총 32억 염기쌍의 0.6%)으로 추정되었다. "기준 인간 게놈에서 410만 570만명의 사이트의 전형적인[개인]게놈 차이[...]시퀀스의 20만개의 염기에 영향을 미치는"Auton A, 브룩스는 LD, 더 번 상원 의원 RM, 개리슨 EP, 강 씨는 HM, 코블 제이미,(알.(10월 2015년)."인간의 유전자 변이에 대한 참고 자료".자연.526년(7571):68–74.Bibcode:2015Natur.526...68T.doi:10.1038/nature15393.PMC 4750478.PMID 26432245.
- ^ "134에 188ka":쿵푸 Q, Mittnik A, 존슨 PL, 소속 K, Lari M, Bollongino R, 태양 C, Giemsch L, 슈미츠 R, 버거 JRonchitelli AM, 마티니 F, Cremonesi RG, Svoboda J, 바우어 P, Caramelli D, 카스텔라노 S, 제국 D, Pääbo S, 크라우제 J(3월 21일 2013년)."인간의 진화를 위해 개정된 시간 고대 미토콘드리아의 게놈에 따라".현재 생물학. 23(7):553–59. doi:10.1016/j.cub.2013.02.044.PMC 5036973.PMID 23523248..
- ^ Patterson N, Richter DJ, Gnerre S, Lander ES, Reich D (2006). "Genetic evidence for complex speciation of humans and chimpanzees". Nature. 441 (7097): 1103–8. Bibcode:2006Natur.441.1103P. doi:10.1038/nature04789. PMID 16710306. S2CID 2325560.
- ^ Jump up to: a b c Chen, F.C.; Li, W.H. (2001). "Genomic divergences between humans and other hominoids and the effective population size of the common ancestor of humans and chimpanzees". Am J Hum Genet. 68 (2): 444–456. doi:10.1086/318206. PMC 1235277. PMID 11170892.
- ^ Kitzmiller 대 Dover 사건의 Ken Miller 사건 재판 기록.
- ^ Sarich, V.M.; Wilson, A.C. (1967). "Immunological time scale for hominid evolution". Science. 158 (3805): 1200–1203. Bibcode:1967Sci...158.1200S. doi:10.1126/science.158.3805.1200. PMID 4964406. S2CID 7349579.
- ^ Venn, Oliver; Turner, Isaac; Mathieson, Iain; de Groot, Natasja; Bontrop, Ronald; McVean, Gil (June 2014). "Strong male bias drives germline mutation in chimpanzees". Science. 344 (6189): 1272–1275. Bibcode:2014Sci...344.1272V. doi:10.1126/science.344.6189.1272. PMC 4746749. PMID 24926018.
- ^ Yoder, A.D.; Yang, Z. (1 July 2000). "Estimation of primate speciation dates using local molecular clocks". Mol Biol Evol. 17 (7): 1081–1090. doi:10.1093/oxfordjournals.molbev.a026389. PMID 10889221.
- ^ Takahata, N.; Satta, Y.; Klein, J. (1995). "Divergence time and population size in the lineage leading to modern humans". Theor Popul Biol. 48 (2): 198–221. doi:10.1006/tpbi.1995.1026. PMID 7482371.
- ^ Yang, Z. (1 December 2002). "Likelihood and Bayes estimation of ancestral population sizes in hominoids using data from multiple loci" (abstract page). Genetics. 162 (4): 1811–1823. doi:10.1093/genetics/162.4.1811. PMC 1462394. PMID 12524351.
- ^ Jump up to: a b c d Chimpanzee Sequencing; Analysis Consortium (2005). "Initial sequence of the chimpanzee genome and comparison with the human genome". Nature. 437 (7055): 69–87. Bibcode:2005Natur.437...69.. doi:10.1038/nature04072. PMID 16136131.
- ^ Olson, M.V. (1999). "When less is more: gene loss as an engine of evolutionary change". Am J Hum Genet. 64 (1): 18–23. doi:10.1086/302219. PMC 1377697. PMID 9915938.
- ^ Jump up to: a b Wang, X.; Grus, W.E.; Zhang, J. (2006). "Gene losses during human origins". PLOS Biol. 4 (3): e52. doi:10.1371/journal.pbio.0040052. PMC 1361800. PMID 16464126.
- ^ Demuth, Jeffery P.; Bie, Tijl De; Stajich, Jason E.; Cristianini, Nello; Hahn, Matthew W. (December 2006). Borevitz, Justin (ed.). "The Evolution of Mammalian Gene Families". PLOS ONE. 1 (1): e85. Bibcode:2006PLoSO...1...85D. doi:10.1371/journal.pone.0000085. PMC 1762380. PMID 17183716.
- ^ Winter, H.; Langbein, L.; Krawczak, M.; Cooper, D.N.; Suarez, L.F.J.; Rogers, M.A.; Praetzel, S.; Heidt, P.J.; Schweizer, J. (2001). "Human type I hair keratin pseudogene phihHaA has functional orthologs in the chimpanzee and gorilla: evidence for recent inactivation of the human gene after the Pan-Homo divergence". Hum Genet. 108 (1): 37–42. doi:10.1007/s004390000439. PMID 11214905. S2CID 21545865.
- ^ Stedman, H.H.; Kozyak, B.W.; Nelson, A.; Thesier, D.M.; Su, L.T.; Low, D.W.; Bridges, C.R.; Shrager, J.B.; Purvis, N.M.; Mitchell, M.A. (2004). "Myosin gene mutation correlates with anatomical changes in the human lineage". Nature. 428 (6981): 415–418. Bibcode:2004Natur.428..415S. doi:10.1038/nature02358. PMID 15042088. S2CID 4304466.
- ^ Perry, G.H.; Verrelli, B.C.; Stone, A.C. (2005). "Comparative analyses reveal a complex history of molecular evolution for human MYH16". Mol Biol Evol. 22 (3): 379–382. doi:10.1093/molbev/msi004. PMID 15470226.
- ^ Hellen, Elizabeth H. B.; Kern, Andrew D. (2015-04-01). "The Role of DNA Insertions in Phenotypic Differentiation between Humans and Other Primates". Genome Biology and Evolution. 7 (4): 1168–1178. doi:10.1093/gbe/evv012. ISSN 1759-6653. PMC 4419785. PMID 25635043.
- ^ Bird, Christine P.; Liu, Maureen; et al. (2007). "Fast-evolving noncoding sequences in the human genome". Genome Biology. 8 (6): R118. doi:10.1186/gb-2007-8-6-r118. PMC 2394770. PMID 17578567.
- ^ Cheng, Z.; Ventura, M.; She, X.; Khaitovich, P.; Graves, T.; Osoegawa, K.; Church, D; Pieter DeJong, P.; Wilson, R. K.; Paabo, S.; Rocchi, M; Eichler, E. E. (2005). "A genome-wide comparison of recent chimpanzee and human segmental duplications". Nature. 437 (1 September 2005): 88–93. Bibcode:2005Natur.437...88C. doi:10.1038/nature04000. PMID 16136132. S2CID 4420359.
- ^ Kaessmann, H.; Heissig, F.; von Haeseler, A.; Pääbo, S. (1999). "DNA sequence variation in a non-coding region of low recombination on the human X chromosome". Nat Genet. 22 (1): 78–81. doi:10.1038/8785. PMID 10319866. S2CID 9153915.
- ^ Saey, Tina Hesman (2009). "Story one: Team decodes neandertal DNA: Genome draft may reveal secrets of human evolution". Science News. 175 (6): 5–7. doi:10.1002/scin.2009.5591750604.
- ^ Green, Richard E.; Krause; Briggs; Maricic; Stenzel; Kircher; Patterson; Li; Zhai; Fritz; Hansen; Durand; Malaspinas; Jensen; Marques-Bonet; Alkan; Prüfer; Meyer; Burbano; Good; Schultz; Aximu-Petri; Butthof; Höber; Höffner; Siegemund; Weihmann; Nusbaum; Lander; et al. (2010-05-07). "A Draft Sequence of the Neandertal Genome". Science. 328 (5979): 710–722. Bibcode:2010Sci...328..710G. doi:10.1126/science.1188021. PMC 5100745. PMID 20448178.
- ^ "NEANDERTALS LIVE!". john hawks weblog. 6 May 2010. Retrieved 2010-12-31.
- ^ Tishkoff, SA.; Reed, FA.; Friedlaender, FR.; Ehret, C.; Ranciaro, A.; Froment, A.; Hirbo, JB.; Awomoyi, AA.; et al. (May 2009). "The genetic structure and history of Africans and African Americans". Science. 324 (5930): 1035–44. Bibcode:2009Sci...324.1035T. doi:10.1126/science.1172257. PMC 2947357. PMID 19407144.
- ^ BBC 월드 뉴스 "아프리카의 유전적 비밀 잠금 해제" 2009년 5월 1일.
- ^ Peng, Y.; et al. (2010). "The ADH1B Arg47His polymorphism in East Asian populations and expansion of rice domestication in history". BMC Evolutionary Biology. 10: 15. doi:10.1186/1471-2148-10-15. PMC 2823730. PMID 20089146.
- ^ 돌연변이의 영향을 받는 특성은 땀샘, 치아, 머리카락 굵기, 유방 조직이다. Kamberov; et al. (2013). "Modeling Recent Human Evolution in Mice by Expression of a Selected EDAR Variant". Cell. 152 (4): 691–702. doi:10.1016/j.cell.2013.01.016. PMC 3575602. PMID 23415220. 2013년 2월 14일 3만 5천년 된 돌연변이와 연관된 동아시아 신체적 특징들
- ^ NCBI (2017-05-08). "dbSNP's human build 150 has doubled the amount of RefSNP records!". NCBI Insights. Retrieved 2017-05-16.
- ^ Jorde, LB; Wooding, SP (2004). "Genetic variation, classification and 'race'". Nature Genetics. 36 (11s): S28–33. doi:10.1038/ng1435. PMID 15508000.
- ^ Tishkoff, SA; Kidd, KK (2004). "Implications of biogeography of human populations for 'race' and medicine". Nature Genetics. 36 (11s): S21–7. doi:10.1038/ng1438. PMID 15507999.
- ^ The 1000 Genomes Project Consortium (2015-10-01). "A global reference for human genetic variation". Nature. 526 (7571): 68–74. Bibcode:2015Natur.526...68T. doi:10.1038/nature15393. ISSN 0028-0836. PMC 4750478. PMID 26432245.
- ^ Mondal, Mayukh; Bertranpedt, Jaume; Leo, Oscar (16 January 2019). "Approximate Bayesian computation with deep learning supports a third archaic introgression in Asia and Oceania". Nature Communications. 10 (246): 246. Bibcode:2019NatCo..10..246M. doi:10.1038/s41467-018-08089-7. PMC 6335398. PMID 30651539.
- ^ Dockrill, Peter (11 February 2019). "Artificial Intelligence Has Found an Unknown 'Ghost' Ancestor in The Human Genome". ScienceAlert.com. Retrieved 11 February 2019.
- ^ Burrell, Teal (29 December 2019). "Scientists Put a Human Intelligence Gene Into a Monkey. Other Scientists are Concerned". Discover. Retrieved 30 December 2019.
- ^ Shi, Lei; et al. (27 March 2019). "Transgenic rhesus monkeys carrying the human MCPH1 gene copies show human-like neoteny of brain development". Chinese National Science Review. 6 (3): 480–493. doi:10.1093/nsr/nwz043. Retrieved 30 December 2019.
추가 읽기
- Jobling, Mark A.; Hollox, Edward; Hurles, Matthew; Kivisild, Toomas; Tyler-Smith, Chris (2013). Human Evolutionary Genetics. New York: Garland Science. ISBN 978-0-8153-4148-2. OCLC 829099073.
- Rannala B, Yang Z (August 2003). "Bayes estimation of species divergence times and ancestral population sizes using DNA sequences from multiple loci". Genetics. 164 (4): 1645–56. doi:10.1093/genetics/164.4.1645. PMC 1462670. PMID 12930768.