숙성탈수소효소
Succinate dehydrogenase| 탈수소효소(탈수소-아미노산산화효소) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
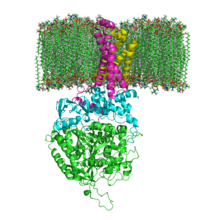 인광 막에 있는 SQR의 구조. SdhA, SdhB, SdhC 및 SdhD | |||||||||
| 식별자 | |||||||||
| EC 번호 | 1.3.5.1 | ||||||||
| CAS 번호. | 9028-11-9 | ||||||||
| 데이터베이스 | |||||||||
| 인텐츠 | IntEnz 뷰 | ||||||||
| 브렌다 | 브렌다 입력 | ||||||||
| 엑스퍼시 | 나이스자이메 뷰 | ||||||||
| 케그 | KEG 입력 | ||||||||
| 메타사이크 | 대사통로 | ||||||||
| 프리암 | 프로필 | ||||||||
| PDB 구조 | RCSB PDB PDBe PDBsum | ||||||||
| 진 온톨로지 | 아미고 / 퀵고 | ||||||||
| |||||||||
| 숙성탈수소효소 | |
|---|---|
| 식별자 | |
| 기호 | 호흡 복합체 II |
| OPM 슈퍼 패밀리 | 3 |
| OPM단백질 | 1조이 |
| 멤브라노메 | 656 |
수진탈수소효소(SDH) 또는 수진-코엔자임 Q 환원효소(SQR) 또는 호흡 복합체 II는 효소 복합체로, 많은 박테리아 세포와 진핵생물의 내측 미토콘드리아 막에서 발견된다. 구연산 사이클과 전자전달 체인에 모두 참여하는 유일한 효소다.[1] 근육에서 높은 숙성 탈수소효소를 보이는 히스토케미컬 분석은 높은 미토콘드리아 함량과 높은 산화 가능성을 보여준다.[2]
구연산 사이클의 6단계에서, SQR은 유비퀴니온을 유비퀴니놀로 환원시켜 후마린산화에 대한 산화 촉매를 한다. 이것은 내측 미토콘드리아 막에서 두 반응을 결합하여 발생한다.
구조
수부닛
미토콘드리아와 많은 박테리아 SQR은 구조적으로 서로 다른 4개의 하위 유니트로 구성되어 있다: 2개의 친수성과 2개의 친수성. 처음 두 서브유닛인 플라보프로테인(SdHA)과 철황단백질(SdhB)은 복합체의 효소 활동이 일어나는 친수성 머리를 형성한다. SdHA에는 균일하게 부착된 플라빈 아데닌 디뉴클레오티드(FAD) 공동 인자 및 숙주 결합 부위가 포함되어 있으며, SdhB에는 [2Fe-2S], [4Fe-4S], [3Fe-4S]의 세 개의 철황 군집이 포함되어 있다. 두 번째 서브유닛은 소수성 멤브레인 앵커 서브유닛, SdhC와 SdhD이다. 인간 미토콘드리아는 SdhA(Fp 서브유닛 타입 I과 타입 II)의 두 가지 뚜렷한 등소형태를 포함하고 있으며, 이러한 등소형태는 아스카리스수움(Ascaris sumum)과 새노하브디트(Caenorhabditis) 선충에서도 발견된다.[3] 서브유닛은 1 헤메 b 그룹과 유비퀴논 결합 부위가 포함된 6개의 트랜섬브레인 나선으로 막 결합 시토크롬 b 콤플렉스를 형성한다. SdhC와 SdhD 서브유닛(이미지에는 표시되지 않음)에서도 두 개의 인광분자, 즉 심도리핀 1개와 인광티딜타놀라민 1개가 발견된다. 그들은 헤메 b 아래의 소수성 공간을 점유하는 역할을 한다. 이러한 서브유닛은 첨부된 영상에 표시된다. SdhA는 녹색, SdhB는 티알, SdhC는 푸치아, SdhD는 노란색이다. SdhC와 SdhD 주위에는 이미지 상단에 중간 공간이 있는 인광질 막이 있다.[4]
단위구성표[5]
| 아니요. | 서브유닛명 | 인간단백질 | 유니프로트의 단백질 설명 | 인간 단백질을 가진 Pfam 계열 |
|---|---|---|---|---|
| 1 | SdhA | SDHA_HUMAN | 숙성탈수소효소 [유비퀴논] 플라보프로테아제 서브유닛, 미토콘드리아 | Pfam PF00890, Pfam PF02910 |
| 2 | SdhB | SDHB_HUMAN | 숙성탈수소효소 [유비퀴논] 철-황산소단위, 미토콘드리아 | Pfam PF13085, Pfam PF13183 |
| 3 | SdhC | C560_HUMAN | 숙성 탈수소효소 시토크롬 b560 서브유닛, 미토콘드리아 | Pfam PF01127 |
| 4 | SDHD | DHSD_HUMAN | 숙성 탈수소효소 [유비퀴논] 시토크롬 b 소단위, 미토콘드리아 | Pfam PF05328 |
유비쿼시노네 바인딩
포유류 SDH에는 매트릭스-최소 Q와P 매트릭스-이상 Q라는D 두 가지 독특한 유비쿼터스니온 결합 사이트가 인식될 수 있다. 유비쿼니콘에 대한 친화력이 더 높은 유비쿼터스니온 결합 사이트 Qp는 SdhB, SdhC, SdhD로 구성된 틈새에 위치한다. 유비퀴논은 서브유닛 B의 His207, 서브유닛 C의 Ser27과 Arg31, 서브유닛 D의 Tyr83의 사이드 체인에 의해 안정화된다. 퀴논 링은 서브 유닛 C의 Ile28과 서브 유닛 B의 Pro160에 둘러싸여 있다. 이러한 잔류물은 서브유닛 B의 Il209, Trp163, Trp164, 서브유닛 C의 Ser27(C원자)과 함께 퀴논 결합형 포켓 Qp의 소수성 환경을 형성한다.[6] 이와는 대조적으로, 메모리 간 공간에 더 가까운 유비퀴논 결합D 사이트 Q는 SdhD로만 구성되며 유비퀴논에 대한 친화력이 낮다.[7]
바인딩 사이트 굴복
SdhA는 숙주의 산화를 위한 결합 부지를 제공한다. 측면 사슬인 Thr254, His354, 아그399 서브유닛 A의 분자를 안정화시키는 한편 FAD는 산화하여 전자를 제1철황 군집인 [2Fe-2S][8]로 운반한다. 이것은 이미지 5에서 볼 수 있다.
리독스 센터
굴복 결합 사이트와 유비쿼시논 결합 사이트는 FAD와 철황 클러스터를 포함한 일련의 리독스 센터로 연결된다. 이 체인은 효소 모노머를 통해 40 40 이상 확장된다. 중심 사이의 모든 에지 대 에지 거리는 생리학적 전자 전달에 대해 제안된 14 å 한계보다 작다.[4] 이 전자전달은 이미지 8에 설명되어 있다.
서브유닛 E
| SdHE | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 NMA1147 단백질의 NMR 구조물을 Neisseria meningitidis에서 추출한다. 동북구조유전체 컨소시엄 대상 mr19 | |||||||||
| 식별자 | |||||||||
| 기호 | SdHE | ||||||||
| Pfam | PF03937 | ||||||||
| 인터프로 | IPR005631 | ||||||||
| |||||||||
분자생물학에서 Sdh5라는 단백질 영역은 또한 SdhE라고 불리며, 이것은 숙성 탈수소효소 단백질 E를 의미한다. 과거에도 ygfY, DUF339라는 이름이 붙었다.[9] SdhE의 또 다른 이름은 숙성 탈수소효소 조립 인자 2(Sdhaf2)이다.[10] 이 단백질은 니세리아 메닌기티디스(NMA1147), 대장균(Eschericia meningitidis)의 YgfY 등 진핵생물과 원핵생물의 보존도가 높은 작은 단백질 그룹에 속한다.[12] SdhE 단백질은 미토콘드리아 막에서 발견된다. 그것은 전자 운송 체인으로 명명된 과정을 통해 에너지를 생성하는데 중요하다.[9]
함수
SdhE의 기능은 숙성 탈수소효소의 향미제로 설명되어 왔다. SdhE는 SdhA에 FAD를 접목한 공동 팩터 샤페론 역할을 한다. 이로 인해 적절한 기능에 필요한 SdhA 플라비닐이 탈수소효소는 탈수소효소다. 연구는 SdhE가 유일한 탄소 공급원으로서 그리고 추가적으로 에너지를 생산하는 전자 운송 체인의 중요한 요소인 숙성 탈수소효소의 기능을 위해 숙성을 위해 박테리아에 의해 요구된다는 것을 보여준다.[9]
구조
이들 단백질의 구조는 5개의 알파헬리크로 구성된 복합다발로 구성되는데, 상향 3헥스 다발과 직교 2헥스 다발로 구성된다.[12]
단백질 상호작용
SDHE는 SDH 복합체의 촉매 서브유닛과 상호작용한다.[13]
인간병
SDH5라는 이름의 인간 유전자는 SdHE 단백질을 위해 암호화된 것이다. 유전자 자체는 염색체 위치 11q13.1에 위치한다. 기능상실 돌연변이는 신경내분비 종양인 파라강리오마를 유발한다.[13]
역사
박테리아 플라비닐에 SdhE가 필요하다는 최근의 연구는 SdhE에 대한 이전의 생각과 모순된다. 원래 FAD를 박테리아 플라보프로테이트에 통합하는 것은 자기분석적인 과정이라고 제안되었다. 최근 연구들은 현재 SdhE가 박테리아에서 플라비닐화에 필요한 것으로 확인된 최초의 단백질이라고 주장한다. 역사적으로 SdhE 단백질은 한때 가상의 단백질로 여겨졌다.[9] ygfY도 전사 규제에 관여하는 것으로 생각됐다.[12]
조립 및 성숙
인간 미토콘드리아 SDH의 모든 하위 단위는 핵 게놈으로 암호화된다. 번역 후 SDHA 서브유닛은 아포프로테인(apoprotein)으로 미토콘드리아 매트릭스로 번역된다. 그 후 첫 번째 단계 중 하나는 FAD 공효소(플라비닐화)의 공밸런스 결합이다. 이 과정은 일부 삼차복시산 사이클 매개체에 의해 규제되는 것으로 보인다. 특히 SDHA의 플라비닐화 자극에 굴복, 이산화물 및 구연산염.[14] eukaryotic Sdh1(포유류의 경우 SDHA)의 경우 FAD 결합 과정에 또 다른 단백질이 필요하다. 즉, 효모의 Sdh5, 포유류 세포의 탈수소효소 결합 인자 2(SDHAF2)이다.
서브유닛 SDHB로 헤테로디머를 형성하기 전에, 공동 결합 FAD를 가진 SDHA의 일부 부분이 다른 조립 요소인 SDHAF4(이스트의 Sdh8)와 상호작용하는 것으로 보인다. Unbound flavorated SDHA는 보호막 역할을 하는 SDHAF4로 디머라이징한다. 연구에 따르면 SDHA-SDHB 조광기의 형성은 SDHAF4가 없을 경우 손상되어 보호자와 유사한 조립 인자가 서브유닛의 상호작용을 촉진할 수 있다. 더욱이 SDHAF4는 결합되지 않은 단수성 SDHA 서브유닛에 의해 여전히 산화될 수 있는 전자들을 수용함으로써 ROS 생성을 방해하는 것으로 보인다.[7]
SDHB의 Fe-S 보형물 그룹은 단백질 복합체 ISU에 의해 미토콘드리아 매트릭스에서 미리 형성되고 있다. 이 단지는 또한 성숙기에 SDHB에 철-설퍼 클러스터를 삽입할 수 있는 것으로 생각된다. 연구는 Fe-S 클러스터 삽입이 SDHA-SDHB 조광기 형성에 선행한다는 것을 시사한다. 그러한 결합을 위해서는 SDHB의 활성 부위 내의 사이스틴 잔류물을 줄여야 한다. 감소된 사이스틴 잔류물과 이미 통합된 Fe-S 클러스터는 모두 ROS 손상에 매우 취약하다. SdHAF1(Sdh6)과 SDHAF3(효모 내 Sdh7) 등 두 가지 SDH 어셈블리 인자는 ROS에 의해 야기되는 Fe-S 클러스터 손상으로부터 서브 유닛 또는 다이머 SDHA-SDHB를 보호하는 방식으로 SDHB 성숙에 관여하는 것으로 보인다.[7]
SDHC와 SDHD로 구성된 소수성 앵커의 조립은 여전히 불분명하다. 특히 heme b 삽입의 경우, 그리고 그 기능까지. Heme b 보형물 그룹은 복합체 II 내에서 전자 운송 경로의 일부로 보이지 않는다.[15] 공동 작용자는 오히려 앵커 안정성을 유지한다.
메커니즘
숙사산화
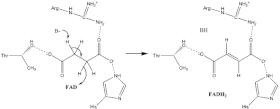
정확한 숙성 산화 메커니즘에 대해서는 거의 알려져 있지 않다. 그러나 결정 구조는 서브유닛 A(표시되지 않음)의 FAD, Glu255, Arg286, His242가 초기 감압 단계에 적합한 후보임을 보여준다. 그 후에 두 가지 가능한 제거 메커니즘이 있다: E2 또는 E1cb. E2 제거에서는 메커니즘이 일치한다. 기본 잔류물 또는 공작용제는 알파 탄소를 감압하고, FAD는 베타 탄소의 하이드라이드를 수용하여 바운드가 후마레이트(fumarate)에 굴복하는 산화를 한다(이미지 6 참조). E1cb에서는 FAD가 하이드라이드를 수용하기 전에 이미지 7에 표시된 에놀레이트 중간이 형성된다. 숙신 탈수소효소에서 어떤 제거 메커니즘이 숙성되는지 결정하기 위해서는 추가 연구가 필요하다. 산화된 후마레이트는 현재 활동 부위와 느슨하게 결합되어 있으며 단백질을 자유롭게 배출할 수 있다.
전자 터널링
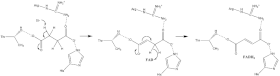
전자가 FAD를 통해 숙성 산화로부터 파생된 후, [3Fe-4S] 클러스터에 도달할 때까지 [Fe-S] 릴레이를 따라 터널링한다. 이 전자들은 이후 활성 부위 내의 대기 중인 유비쿼시논 분자로 전달된다. Iron-Sulfur 전자 터널링 시스템은 이미지 9에 나타나 있다.
유비쿼시논 감소
유비쿼시논의 O1 카보닐 산소는 서브유닛 D의 Tyr83과의 수소 결합 상호작용에 의해 활성 부위(이미지 4)를 향한다. [3Fe-4S] 철황 성단에 전자가 존재하면 유비퀴논이 제2의 방향으로 이동하게 된다. 이를 통해 유비쿼시논의 O4 카보닐 그룹과 서브유닛 C의 Ser27 사이의 두 번째 수소 결합 상호작용을 촉진한다. 첫 번째 단일 전자 감소 단계에 이어, 세미키논 급진 종(種)이 형성된다. 두 번째 전자는 유비퀴니온을 유비퀴니놀로 완전히 감소시키기 위해 [3Fe-4S] 군집으로부터 도착한다. 유비퀴니온 감소의 이 메커니즘은 이미지 8에 나타나 있다.
헤메 보형물군
비록 숙성된 탈수소효소에 있는 heme의 기능은 여전히 연구되고 있지만[by whom?], 일부 연구는 [3Fe-4S]를 통해 유비쿼시논에 전달된 첫 번째 전자가 heme와 유비쿼시논 중간값 사이를 앞뒤로 터널링할 수 있다고 주장했다. 이렇게 해서 헤메 공효소는 전자 싱크대 역할을 한다. 그 역할은 분자 산소와 중간이 상호작용을 하여 반응산소종(ROS)을 생산하는 것을 막는 것이다. 유비쿼시논에 상대적인 헤메 그룹은 이미지 4에 나타나 있다.
또한 전자가 [3Fe-4S] 군집으로부터 heme에 직접 터널링하는 것을 방지하기 위한 탕구 메커니즘이 있을 수 있다고 제안되었다. 잠재적 후보로는 His207이 남아 있는데, 이것은 성단과 heme 사이에 바로 놓여 있다. 서브유닛 B의 His207은 [3Fe-4S] 군집, 바운드 유비쿼니온 및 헤메와 직접 인접하며, 이러한 레독스 중심 사이의 전자 흐름을 변조할 수 있다.[16]
양성자 전이
SQR에서 퀴논을 완전히 줄이기 위해서는 두 개의 양성자뿐만 아니라 두 개의 전자가 필요하다. 물 분자(HOH39)가 활성 현장에 도착하여 서브유닛 B의 His207, 서브유닛 C의 Arg31, 서브유닛 D의 Asp82에 의해 조정된다는 주장이 제기되었다. 세미키논 종은 HOH39에서 전달된 양성자에 의해 양성되어 유비퀴논을 유비퀴니놀로 환원시키는 것을 완성한다. His207과 Asp82는 이 과정을 용이하게 할 것이다. 다른 연구들은 서브유닛 D의 Tyr83이 유비쿼시논의 O1 카보닐 산소뿐만 아니라 가까운 히스티딘에 조정된다고 주장한다. 히스티딘 잔여물은 티로신(tyrosine)의 pKa를 감소시켜 양성자를 환원된 유비쿼시논 중간재에 기부하는 것이 더 적합하다.
억제제
복합 II에는 두 가지 뚜렷한 등급의 억제제(SDHI)가 있다. 즉, 굴복한 주머니에서 결합되는 억제제와 유비쿼니온 주머니에서 결합되는 억제제다. 유비퀴논 타입 억제제로는 카르복신과 디오일트리플루오로아세톤이 있다. 숙신-아날로그 억제제에는 합성 화합물 말론산염뿐만 아니라 TCA 사이클 매개체, 말산염 및 옥살로아세테이트도 포함된다. 실제로 옥살로아세테이트는 콤플렉스 II의 가장 강력한 억제제 중 하나이다. 공통의 TCA 사이클 중간이 콤플렉스 II를 억제하는 이유는 완전히 이해되지는 않지만, 콤플렉스 I에 의해 과산화수소의 역전자 전달 매개 생산을 최소화하는데 보호적 역할을 할 수 있다.[17] 앳페닌 5a는 유비쿼시논 결합을 모방한 강력한 콤플렉스 II 억제제다.
유비퀴논 타입 억제제는 1960년대 이후 농업에서 살균제로 사용되어 왔다. 카르복신은 주로 줄기 녹, 리옥토니아병 등 기저귀근육종(basidiodomycetes)에 의한 질병을 다스리는 데 사용되었다. 보다 최근에, 다양한 식물 병원균에 대한 스펙트럼이 넓은 다른 화합물들이 개발되었다. 보스카리드, 펜티호피라드, 플루오피람.[18] 일부 농업적으로 중요한 균류는 새로운 세대의 유비쿼시논 타입 억제제의 구성원에 민감하지 않다.
질병에서의 역할
미토콘드리아의 전자전달 체인에 있어서 숙성-코엔자임 Q 환원효소의 근본적인 역할은 그것을 대부분의 다세포 유기체에서 중요하게 만들고, 게놈에서 이 효소를 제거하는 것은 쥐의 배아 단계에서도 치명적인 것으로 나타났다.
- SdhA 돌연변이는 레이 증후군, 미토콘드리아 뇌병증, 시신경 위축으로 이어질 수 있다.
- sdhB 돌연변이는 크로마핀 세포에서 종양유전증을 유발할 수 있으며, 유전성 파라강피종과 유전성 페로모세포종 등 숙성 탈수소효소 결핍 종양, 탈수소효소 결핍성 위장성 기형종양(GIST)을 유발할 수 있다.[20] 종양은 악성인 경향이 있다. 그것은 또한 수명을 줄이고 과산화수소 이온의 생산을 증가시킬 수 있다.
- SdhC 돌연변이는 수명의 감소, 과산화이온의 생산량 증가, 유전성 파라강리종, 유전성 페로크로모시토종으로 이어질 수 있다. 종양은 양성인 경향이 있다. 이런 돌연변이는 흔치 않다.
- sdhD 돌연변이는 유전성 파라강리종과 유전성 페로크로모사이토종으로 이어질 수 있다. 종양은 양성인 경향이 있으며, 머리와 목 부위에 자주 발생한다. 이러한 돌연변이는 또한 수명을 줄이고 과산화수소 이온의 생산을 증가시킬 수 있다.
포유류는 미토콘드리아 에너지 생성에서뿐만 아니라 산소 감지 및 종양 억제에도 역할을 하므로 현재 진행 중인 연구의 대상이다.
복합 II의 주성분인 미토콘드리아 효소 숙성탈수소효소(SDH)의 감소된 수치는 헌팅턴병 환자의 뇌에서 사후 모템이 관찰되며, 에너지 대사 결함은 사전자율성 HD 환자와 증상성 HD 환자 모두에서 확인되었다.[21]
참고 항목
참조
- ^ Oyedotun KS, Lemire BD (March 2004). "The quaternary structure of the Saccharomyces cerevisiae succinate dehydrogenase. Homology modeling, cofactor docking, and molecular dynamics simulation studies". The Journal of Biological Chemistry. 279 (10): 9424–31. doi:10.1074/jbc.M311876200. PMID 14672929.
- ^ webmaster (2009-03-04). "Using Histochemistry to Determine Muscle Properties". Succinate Dehydrogenase: Identifying Oxidative Potential. University of California, San Diego. Retrieved 2017-12-27.
- ^ Tomitsuka E, Hirawake H, Goto Y, Taniwaki M, Harada S, Kita K (August 2003). "Direct evidence for two distinct forms of the flavoprotein subunit of human mitochondrial complex II (succinate-ubiquinone reductase)". Journal of Biochemistry. 134 (2): 191–5. doi:10.1093/jb/mvg144. PMID 12966066.
- ^ a b Yankovskaya V, Horsefield R, Törnroth S, Luna-Chavez C, Miyoshi H, Léger C, et al. (January 2003). "Architecture of succinate dehydrogenase and reactive oxygen species generation". Science. 299 (5607): 700–4. Bibcode:2003Sci...299..700Y. doi:10.1126/science.1079605. PMID 12560550. S2CID 29222766.
- ^ Sun F, Huo X, Zhai Y, Wang A, Xu J, Su D, et al. (July 2005). "Crystal structure of mitochondrial respiratory membrane protein complex II". Cell. 121 (7): 1043–57. doi:10.1016/j.cell.2005.05.025. PMID 15989954.
- ^ Horsefield R, Yankovskaya V, Sexton G, Whittingham W, Shiomi K, Omura S, et al. (March 2006). "Structural and computational analysis of the quinone-binding site of complex II (succinate-ubiquinone oxidoreductase): a mechanism of electron transfer and proton conduction during ubiquinone reduction". The Journal of Biological Chemistry. 281 (11): 7309–16. doi:10.1074/jbc.M508173200. PMID 16407191.
- ^ a b c Van Vranken JG, Na U, Winge DR, Rutter J (December 2014). "Protein-mediated assembly of succinate dehydrogenase and its cofactors". Critical Reviews in Biochemistry and Molecular Biology. 50 (2): 168–80. doi:10.3109/10409238.2014.990556. PMC 4653115. PMID 25488574.
- ^ Kenney WC (April 1975). "The reaction of N-ethylmaleimide at the active site of succinate dehydrogenase". The Journal of Biological Chemistry. 250 (8): 3089–94. doi:10.1016/S0021-9258(19)41598-6. PMID 235539.
- ^ a b c d McNeil MB, Clulow JS, Wilf NM, Salmond GP, Fineran PC (2012). "SdhE is a conserved protein required for flavinylation of succinate dehydrogenase in bacteria". J Biol Chem. 287 (22): 18418–28. doi:10.1074/jbc.M111.293803. PMC 3365757. PMID 22474332.
- ^ "SDHAF2 Gene - GeneCards SDHF2 Protein SDHF2 Antibody". Archived from the original on 2011-11-05.
- ^ Liu G, Sukumaran DK, Xu D, Chiang Y, Acton T, Goldsmith-Fischman S, Honig B, Montelione GT, Szyperski T (May 2004). "NMR structure of the hypothetical protein NMA1147 from Neisseria meningitidis reveals a distinct 5-helix bundle". Proteins. 55 (3): 756–8. doi:10.1002/prot.20009. PMID 15103637.
- ^ a b c Lim K, Doseeva V, Demirkan ES, Pullalarevu S, Krajewski W, Galkin A, Howard A, Herzberg O (February 2005). "Crystal structure of the YgfY from Escherichia coli, a protein that may be involved in transcriptional regulation". Proteins. 58 (3): 759–63. doi:10.1002/prot.20337. PMID 15593094.
- ^ a b Hao HX, Khalimonchuk O, Schraders M, Dephoure N, Bayley JP, Kunst H, et al. (August 2009). "SDH5, a gene required for flavination of succinate dehydrogenase, is mutated in paraganglioma". Science. 325 (5944): 1139–42. Bibcode:2009Sci...325.1139H. doi:10.1126/science.1175689. PMC 3881419. PMID 19628817.
- ^ Brandsch R, Bichler V (June 1989). "Covalent cofactor binding to flavoenzymes requires specific effectors". European Journal of Biochemistry. 182 (1): 125–8. doi:10.1111/j.1432-1033.1989.tb14808.x. PMID 2659351.
- ^ Sun F, Huo X, Zhai Y, Wang A, Xu J, Su D, et al. (July 2005). "Crystal structure of mitochondrial respiratory membrane protein complex II". Cell. 121 (7): 1043–57. doi:10.1016/j.cell.2005.05.025. PMID 15989954.
- ^ Tran QM, Rothery RA, Maklashina E, Cecchini G, Weiner JH (October 2006). "The quinone binding site in Escherichia coli succinate dehydrogenase is required for electron transfer to the heme b". The Journal of Biological Chemistry. 281 (43): 32310–7. doi:10.1074/jbc.M607476200. PMID 16950775.
- ^ Muller FL, Liu Y, Abdul-Ghani MA, Lustgarten MS, Bhattacharya A, Jang YC, Van Remmen H (January 2008). "High rates of superoxide production in skeletal-muscle mitochondria respiring on both complex I- and complex II-linked substrates". The Biochemical Journal. 409 (2): 491–9. doi:10.1042/BJ20071162. PMID 17916065.
- ^ Avenot HF, Michailides TJ (2010). "Progress in understanding molecular mechanisms and evolution of resistance to succinate dehydrogenase inhibiting (SDHI) fungicides in phytopathogenic fungi". Crop Protection. 29 (7): 643–651. doi:10.1016/j.cropro.2010.02.019.
- ^ Dubos T, Pasquali M, Pogoda F, Casanova A, Hoffmann L, Beyer M (January 2013). "Differences between the succinate dehydrogenase sequences of isopyrazam sensitive Zymoseptoria tritici and insensitive Fusarium graminearum strains". Pesticide Biochemistry and Physiology. 105 (1): 28–35. doi:10.1016/j.pestbp.2012.11.004. PMID 24238287.
- ^ Barletta JA, Hornick JL (July 2012). "Succinate dehydrogenase-deficient tumors: diagnostic advances and clinical implications". Advances in Anatomic Pathology. 19 (4): 193–203. doi:10.1097/PAP.0b013e31825c6bc6. PMID 22692282. S2CID 32088940.
- ^ Skillings EA, Morton AJ (2016). "Delayed Onset and Reduced Cognitive Deficits through Pre-Conditioning with 3-Nitropropionic Acid is Dependent on Sex and CAG Repeat Length in the R6/2 Mouse Model of Huntington's Disease". Journal of Huntington's Disease. 5 (1): 19–32. doi:10.3233/JHD-160189. PMID 27031731.