DNA 복구
DNA repair
DNA 복구는 세포가 유전체를 암호화하는 DNA 분자의 손상을 확인하고 수정하는 과정의 집합입니다.[1] 인간 세포에서는 정상적인 대사 활동과 방사선과 같은 환경적 요인 모두 DNA 손상을 일으켜 하루에 세포당 수만 개의 개별 분자 병변이 발생할 수 있습니다.[2] 이러한 병변의 대부분은 DNA 분자에 구조적 손상을 일으키고 영향을 받은 DNA가 암호화하는 유전자를 전사하는 세포의 능력을 변경하거나 제거할 수 있습니다. 다른 병변은 세포의 유전체에 잠재적으로 해로운 돌연변이를 유발하고, 이는 유사분열을 겪은 후 딸 세포의 생존에 영향을 미칩니다. 그 결과 DNA 구조의 손상에 대한 반응으로 DNA 복구 과정이 끊임없이 활성화됩니다. 정상적인 복구 과정이 실패했을 때, 그리고 세포 사멸이 일어나지 않았을 때 회복할 수 없는 DNA 손상이 발생할 수 있습니다. 이는 결국 두 개의 히트 가설에 따라 악성 종양, 즉 암으로 이어질 수 있습니다.
DNA 복구 속도는 세포 유형, 세포의 나이, 세포외 환경 등 많은 요인에 따라 달라집니다. 대량의 DNA 손상이 축적된 세포, 또는 더 이상 DNA에 발생한 손상을 효과적으로 복구하지 않는 세포는 가능한 세 가지 상태 중 하나에 들어갈 수 있습니다.
- 노쇠로 알려진 돌이킬 수 없는 휴면 상태
- 세포 자살, 세포 사멸 또는 프로그램된 세포 사멸로도 알려져 있습니다.
- 조절되지 않는 세포 분열로 인해 암이 발생하는 종양이 형성될 수 있습니다.
세포의 DNA 복구 능력은 유전체의 완전성과 유기체의 정상적인 기능에 필수적입니다. 초기에 수명에 영향을 미치는 것으로 나타났던 많은 유전자들이 DNA 손상 복구와 보호에 관여하는 것으로 밝혀졌습니다.[3]
2015년 노벨 화학상은 DNA 복구 과정의 분자 메커니즘에 대한 연구로 토마스 린달, 폴 모드리치, 아지즈 싼카에게 돌아갔습니다.[4][5]
DNA손상
환경적 요인과 세포 내부의 정상적인 대사 과정으로 인한 DNA 손상은 하루에 세포당 10,000~1,000,000개의 분자 병변이 발생합니다.[2] 이것은 인간 게놈의 약 32억개 염기의 최대 0.0003125%에 불과하지만, 중요한 유전자(종양 억제 유전자 등)의 복구되지 않은 병변은 세포의 기능 수행 능력을 방해하고 종양 형성 가능성을 현저히 증가시키고 종양 이질성에 기여할 수 있습니다.
이중나선의 1차 구조는 DNA 손상의 대부분을 차지하는데, 즉 염기 자체가 화학적으로 변형된 것입니다. 이러한 변형은 다시 표준 이중나선에 맞지 않는 비-천연 화학 결합 또는 부피가 큰 부가물을 도입함으로써 분자의 규칙적인 나선 구조를 파괴할 수 있습니다. 단백질이나 RNA와 달리 DNA는 보통 3차 구조가 부족하기 때문에 그 수준에서는 손상이나 교란이 일어나지 않습니다. 그러나 DNA는 (진핵생물에서) 히스톤이라고 불리는 "포장" 단백질에 매우 많이 감겨 있고 감겨 있으며, 두 상부 구조 모두 DNA 손상의 영향에 취약합니다.
원천
DNA 손상은 크게 두 가지 유형으로 나눌 수 있습니다.
- 정상적인 대사 부산물(spont성 돌연변이)로부터 생성된 활성 산소 종에 의한 공격과 같은 내인성 손상, 특히 산화적 탈아미노화 과정
- 복제 오류도 포함됩니다.
- 등의 외부 작용에 의한 외생적 피해
세포 분열 전에 손상된 DNA가 복제되면 손상된 DNA와 반대되는 잘못된 염기가 통합될 수 있습니다. 이러한 잘못된 염기를 유전하는 딸 세포는 원래의 DNA 염기서열을 복구할 수 없는 돌연변이를 가지고 있습니다(예를 들어 유전자 전환을 통한 등돌연변이의 드문 경우는 제외).
종류들
내인성 세포 과정으로 인한 DNA 손상에는 다음과 같은 몇 가지 유형이 있습니다.
- 염기[예: 8-oxo-7,8-dihydroguanine (8-oxoG)]의 산화 및 활성 산소 종으로부터의 DNA 가닥 중단의 생성,
- 염기의 알킬화(보통 메틸화), 예를 들어 7-메틸구아노신, 1-메틸아데닌, 6-O-메틸구아닌의 형성)
- 탈아미노화, 탈퓨리화 및 탈피리미딘화와 같은 염기의 가수분해.
- "벌키한 부가물 형성"(예: 벤조[a]피렌 디올 에폭사이드-dG 부가물, 아리스톨락탐 I-dA 부가물)
- DNA 복제의 오류로 인해 염기가 일치하지 않거나 잘못된 DNA 염기가 새로 형성되는 DNA 가닥에 제자리에 연결되거나 DNA 염기가 건너뛴다거나 잘못 삽입됩니다.
- DNA 단일 질소염기의 변화로 인한 단일가산물 손상
- 다이덕트손상
외인성 작용제에 의한 손상은 여러 형태로 나타납니다. 몇 가지 예는 다음과 같습니다.
- UV-B 빛은 인접한 시토신과 티민 염기 사이의 가교를 일으켜 피리미딘 이량체를 생성합니다. 이것을 직접적인 DNA 손상이라고 합니다.
- UV-A 빛은 대부분 활성산소를 생성합니다. 활성산소에 의한 손상을 간접 DNA 손상이라고 합니다.
- 방사성 붕괴나 우주선에 의해 생성된 것과 같은 이온화 방사선은 DNA 가닥을 끊어지게 합니다. 중간 수준의 이온화 방사선은 회복할 수 없는 DNA 손상(신생물에 필요한 복제 및 전사 오류를 초래하거나 바이러스 상호작용을 유발할 수 있음)을 유발하여 조기 성숙 노화 및 암을 유발할 수 있습니다.
- 높은 온도에서 열 파괴는 탈퓨린화(DNA backbone에서 퓨린 염기의 손실)와 단일 가닥 파손의 속도를 증가시킵니다. 예를 들어 40~80℃의 온천에서 자라는 호열성 박테리아에서 가수분해성 탈염이 관찰됩니다.[7][8] 이 종들은 탈퓨린화 속도(세대당 유전체당 300개의 퓨린 잔기)가 너무 높아 정상적인 복구 기계로 복구할 수 없기 때문에 적응 반응의 가능성을 배제할 수 없습니다.
- 염화비닐, 과산화수소와 같은 산업용 화학물질과 연기, 그을음, 타르에서 발견되는 다환 방향족 탄화수소와 같은 환경용 화학물질은 매우 다양한 DNA 부가물을 만들어냅니다. - 에탄산염, 산화염기, 알킬화 포스포디에스테르, DNA의 가교.
UV 손상, 알킬화/메틸화, X선 손상 및 산화 손상이 유발 손상의 예입니다. 자발적인 손상에는 염기의 손실, 탈아민, 당 고리 퍼커링 및 자동 이동이 포함될 수 있습니다. 내인성 산화제에 의한 구성적(자발적) DNA 손상은 미처리 세포에서 낮은 수준의 히스톤 H2AX 인산화로 검출될 수 있습니다.[9]
핵 대 미토콘드리아
인간 세포, 그리고 일반적으로 진핵 세포에서 DNA는 핵 안과 미토콘드리아 안의 두 세포 위치에서 발견됩니다. 핵 DNA(n-DNA)는 세포 주기의 비복제 단계에서 염색질로 존재하고 세포 분열 동안 염색체로 알려진 집합 구조로 응축됩니다. 어느 상태에서든 DNA는 매우 압축되어 히스톤이라고 불리는 구슬과 같은 단백질 주위에 감겨 있습니다. 세포가 n-DNA에 암호화된 유전 정보를 발현해야 할 때마다 필요한 염색체 영역이 풀리고, 그 영역에 위치한 유전자가 발현되고, 그 영역이 다시 휴지기 형태로 응축됩니다. 미토콘드리아 DNA(mtDNA)는 미토콘드리아 소기관 내부에 위치하며 여러 개의 사본으로 존재하며 또한 많은 단백질과 밀접하게 연관되어 핵양체로 알려진 복합체를 형성합니다. 산화적 인산화를 통한 아데노신 삼인산(ATP)의 지속적인 생성의 부산물인 활성산소(ROS)는 미토콘드리아 내부에 mtDNA를 손상시키는 것으로 알려진 고도의 산화 환경을 형성합니다. 이 종들의 독성에 대항하는 중요한 효소는 진핵 세포의 미토콘드리아와 세포질 모두에 존재하는 superoxide dismutase입니다.
노쇠와 세포 사멸
세포가 더 이상 분열하지 않는 비가역적 과정인 노화는 텔로미어라고 불리는 염색체 말단의 단축에 대한 보호 반응입니다. 텔로미어는 염색체를 덮고 세포가 분열될 때마다 부분적으로 분해되는 반복적인 비암호화 DNA의 긴 영역입니다(Hayflick limit 참조).[10] 대조적으로, 정지는 유전체 손상과 관련이 없는 가역적인 세포 휴면 상태입니다(세포 주기 참조). 세포의 노화는 공간적인 이유로 세포의 물리적 존재가 유기체에 의해 요구되는 경우 세포자멸사의 기능적 대안으로 작용할 수 있으며,[11] 이는 성장 촉진 세포 신호가 없는 상태에서 DNA가 손상된 세포가 부적절하게 복제되는 것을 방지하는 "최후의 수단" 메커니즘의 역할을 합니다. 조절되지 않는 세포 분열은 유기체에 잠재적으로 치명적인 종양(암 참조)의 형성으로 이어질 수 있습니다. 따라서 노화와 세포 사멸의 유도는 암을 예방하기 위한 전략의 일환으로 여겨집니다.[12]
돌연변이
DNA 손상과 돌연변이를 구분하는 것이 중요한데, 이것은 DNA의 두 가지 주요한 오류입니다. DNA 손상과 돌연변이는 근본적으로 다릅니다. 손상은 DNA에 단일 가닥 및 이중 가닥 파손, 8-하이드록시데옥시구아노신 잔기 및 다환 방향족 탄화수소 부가물과 같은 물리적 이상을 초래합니다. DNA 손상은 효소에 의해 인식될 수 있으므로 상보적인 DNA 가닥 또는 상동 염색체의 손상되지 않은 서열과 같은 중복 정보가 복사에 사용될 수 있는 경우 올바르게 복구될 수 있습니다. 세포가 DNA 손상을 유지하면 유전자의 전사를 막을 수 있으므로 단백질로의 번역도 차단됩니다. 복제가 차단되거나 세포가 죽을 수도 있습니다.
DNA 손상과 대조적으로 돌연변이는 DNA의 염기서열의 변화입니다. 염기 변화가 두 DNA 가닥에 모두 존재하면 효소는 돌연변이를 인식할 수 없으므로 돌연변이를 복구할 수 없습니다. 세포 수준에서 돌연변이는 단백질 기능과 조절에 변화를 일으킬 수 있습니다. 돌연변이는 세포가 복제될 때 복제됩니다. 세포 집단에서 돌연변이 세포는 세포의 생존 및 번식 능력에 대한 돌연변이의 영향에 따라 빈도가 증가하거나 감소할 것입니다.
서로 확연히 다르지만, DNA 손상과 돌연변이는 DNA 손상이 복제나 복구 과정에서 DNA 합성의 오류를 일으키는 경우가 많기 때문에, 이러한 오류는 돌연변이의 주요 원인입니다.
이러한 DNA 손상과 돌연변이의 특성을 볼 때 DNA 손상은 세포를 분할하지 않거나 느리게 분할하는 데 있어 특별한 문제임을 알 수 있으며, 여기서 복구되지 않은 손상은 시간이 지남에 따라 누적되는 경향이 있습니다. 반면 빠르게 분열하는 세포에서는 복제를 차단해 세포를 죽이지 않는 복구되지 않은 DNA 손상이 복제 오류를 일으켜 돌연변이를 일으키는 경향이 있습니다. 중성이 아닌 돌연변이의 대부분은 세포의 생존에 해롭습니다. 따라서, 복제 세포가 있는 조직을 구성하는 세포 집단에서 돌연변이 세포는 손실되는 경향이 있습니다. 그러나 생존 이점을 제공하는 드문 돌연변이는 조직의 이웃 세포를 희생시키면서 클론적으로 확장되는 경향이 있습니다. 이러한 돌연변이 세포가 암을 일으킬 수 있기 때문에 세포에 대한 이러한 이점은 유기체 전체에 불리합니다. 따라서 자주 분열하는 세포의 DNA 손상은 돌연변이를 일으키기 때문에 암의 주요 원인이 됩니다. 대조적으로, 드물게 분열하는 세포의 DNA 손상은 노화의 주요 원인일 가능성이 높습니다.[13]
매커니즘
DNA 손상이 유전체의 필수 정보의 완전성과 접근성을 손상시키면 세포는 기능할 수 없습니다(그러나 세포는 필수적이지 않은 유전자가 없거나 손상되었을 때 표면적으로 기능을 유지합니다). DNA의 이중 나선 구조에 가해지는 손상의 종류에 따라, 잃어버린 정보를 복구하기 위한 다양한 복구 방법이 개발되었습니다. 가능하면 세포는 DNA의 변형되지 않은 상보적 가닥이나 자매 염색질을 주형으로 사용하여 원래 정보를 복구합니다. 템플릿에 액세스하지 않으면 세포는 트랜스레젼 합성으로 알려진 오류가 발생하기 쉬운 복구 메커니즘을 마지막 수단으로 사용합니다.
DNA의 손상은 나선의 공간적 구성을 변화시키며, 그러한 변화는 세포에 의해 감지될 수 있습니다. 손상이 국소화되면 특정 DNA 복구 분자가 손상 부위 또는 근처에서 결합하여 다른 분자가 결합하여 실제 복구가 가능한 복합체를 형성하도록 유도합니다.
직접반전
세포는 DNA를 화학적으로 역전시킴으로써 DNA에 손상을 주는 세 가지 유형을 제거하는 것으로 알려져 있습니다. 이러한 메커니즘은 템플릿이 필요하지 않습니다. 왜냐하면 이러한 메커니즘이 대응하는 손상 유형은 4개의 베이스 중 하나에서만 발생할 수 있기 때문입니다. 이러한 직접적인 반전 메커니즘은 발생한 손상의 유형에 따라 다르며 포스포다이에스터 백본의 파손을 포함하지 않습니다. UV 광 조사 시 피리미딘 이량체의 형성은 인접한 피리미딘 염기 사이에 비정상적인 공유 결합을 초래합니다. 광활성화 과정은 효소 광분해효소의 작용에 의해 이러한 손상을 직접적으로 역전시킵니다. 광활성화는 청색/자외선(300~500nm 파장)에서 흡수된 에너지에 절대적으로 의존하여 촉매 작용을 촉진합니다.[14] 박테리아, 곰팡이, 그리고 대부분의 동물에 존재하는 오래된 효소인 광분해효소는 인간에게는 더 이상 기능을 하지 [15]못하고 대신 뉴클레오티드의 절제를 통해 자외선 조사로 인한 손상을 복구합니다. 또 다른 유형의 손상인 구아닌 염기의 메틸화는 효소 메틸 구아닌 메틸 전이효소(MGMT)에 의해 직접적으로 역전되며, 그 박테리아 등가물을 ogt라고 합니다. 이는 각 MGMT 분자가 한 번만 사용할 수 있기 때문에 비용이 많이 드는 공정입니다. 즉, 반응은 촉매 반응이 아니라 화학량론적입니다.[16] 박테리아의 메틸화제에 대한 일반적인 반응은 적응 반응으로 알려져 있으며 알킬화 복구 효소의 상향 조절에 의해 지속적인 노출 시 알킬화제에 대한 내성 수준을 부여합니다.[17] 세포에 의해 역전된 세 번째 DNA 손상 유형은 염기 시토신과 아데닌의 특정 메틸화입니다.
단일가닥손상

이중나선의 두 가닥 중 한 가닥에만 결함이 있을 때 나머지 한 가닥은 손상된 가닥의 교정을 안내하는 템플릿으로 사용할 수 있습니다. DNA의 두 쌍의 분자 중 하나의 손상을 복구하기 위해 손상된 뉴클레오타이드를 제거하고 손상되지 않은 DNA 가닥에서 발견되는 것과 상보적인 손상되지 않은 뉴클레오타이드로 대체하는 절제 복구 메커니즘이 여러 가지 존재합니다.[16]
- BER(base excision repair): 손상된 단일 염기 또는 뉴클레오티드는 가장 일반적으로 염기 또는 뉴클레오티드를 제거한 후 올바른 염기 또는 뉴클레오티드를 삽입하여 복구됩니다. 글리코실라제[18] 효소는 염기의 절단을 통해 DNA에서 손상된 염기를 제거하고, 염기와 디옥시리보스의 결합을 절단합니다. 이 효소들은 단일 염기를 제거하여 아푸린산 또는 아피리미딘산 부위(AP site)를 만듭니다.[18] AP 엔도뉴클레아제라고 불리는 효소들은 손상된 DNA 골격을 AP 부위에서 긁어냅니다. 그런 다음 DNA 중합효소는 5'에서 3' 사이의 엑소뉴클레아제 활성을 이용하여 손상된 부위를 제거하고 상보적 가닥을 주형으로 하여 새로운 가닥을 정확하게 합성합니다.[18] 그런 다음 효소 DNA 연결효소에 의해 간격이 밀봉됩니다.[19]
- 뉴클레오티드 절제 복구(NER): UV 광에 의한 피리미딘 이량체화와 같은 부피가 크고 나선을 왜곡시키는 손상은 일반적으로 3단계 과정으로 복구됩니다. 먼저 손상이 인식되면 엔도뉴클레아제에 의해 손상 부위의 상류와 하류 모두에서 12-24개의 뉴클레오타이드 길이의 DNA가 제거되고, 제거된 DNA 영역이 다시 합성됩니다.[20] NER은 고도로 진화적으로 보존된 복구 메커니즘으로 거의 모든 진핵세포와 원핵세포에서 사용됩니다.[20] 원핵생물에서 NER은 Uvr 단백질에 의해 매개됩니다.[20] 일반적인 전략은 동일하지만 진핵생물에서는 더 많은 단백질이 관여합니다.[20]
- 교정에 의해 수정되지 않는 오류를 수정하기 위해 모든 셀에 기본적으로 불일치 복구 시스템이 있습니다. 이 시스템은 적어도 두 개의 단백질로 구성됩니다. 하나는 불일치를 감지하고, 다른 하나는 새로 합성된 DNA 가닥을 손상 부위에 가깝게 절단하는 엔도뉴클레아제를 모집합니다. 대장균에서 관련된 단백질은 Mut 클래스 단백질입니다. MutS, MutL, MutH. 대부분의 진핵생물에서 MutS의 유사체는 MSH이고 MutL의 유사체는 MLH입니다. MutH는 세균에만 존재합니다. 이어서 엑소뉴클레아제에 의한 손상된 부위의 제거, DNA 중합효소에 의한 재합성, DNA 연결효소에 의한 닉 밀봉이 진행됩니다.[21]
이중 가닥 파손
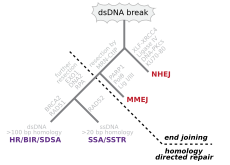
이중나선의 양쪽 가닥이 잘리는 이중가닥 끊어짐은 유전체 재배열을 초래할 수 있기 때문에 세포에 특히 위험합니다. 사실, 두 가닥을 같은 지점에서 연결하는 가교가 이중 가닥 분열을 동반할 때, 두 가닥 모두 복구 메커니즘의 주형으로 사용될 수 없기 때문에 세포가 다음에 분열할 때 유사분열을 완료하지 못하고 죽거나 드물게 돌연변이를 겪게 됩니다.[22][23] 이중 가닥 파손(DSB)을 복구하기 위한 세 가지 메커니즘이 존재합니다: 비-상동 말단 접합(NHEJ), 미세 상동 매개 말단 접합(MMeJ) 및 상동 재조합(HR).[16][24]

- NHEJ에서는 보조인자 XRCC4와 복합체를 형성하는 전문 DNA 연결효소인 DNA 연결효소 IV가 양 말단에 직접 결합합니다.[25] 정확한 복구를 안내하기 위해 NHEJ는 연결될 DNA의 단일 가닥 꼬리에 존재하는 마이크로호몰로지라고 불리는 짧은 상동 서열에 의존합니다. 이러한 돌출부가 호환된다면 일반적으로 수리가 정확합니다.[26][27][28][29] NHEJ는 또한 복구 중에 돌연변이를 일으킬 수 있습니다. 파손 부위에서 손상된 뉴클레오티드가 손실되면 결손이 발생할 수 있으며, 일치하지 않는 말단의 결합은 삽입 또는 전위를 형성합니다. NHEJ는 상동 재조합에 의해 복구할 수 있는 템플릿이 없기 때문에 세포가 DNA를 복제하기 전에 특히 중요합니다. 고등 진핵생물에는 "백업" NHEJ 경로가 있습니다.[30] NHEJ는 유전체 관리자로서의 역할 외에도 척추동물 면역체계에서 B세포와 T세포 수용체에 다양성을 생성하는 과정인 V(D)J 재조합 동안 유도된 헤어핀으로 덮인 이중 가닥 분열을 결합하는 데 필요합니다.[31]
- MMEJ는 이중 가닥 절단의 양쪽에서 MRE11 뉴클레아제에 의한 근거리 말단 절제로 시작하여 미세 상동성 영역을 드러냅니다.[32] 추가 단계에서,[33] Poly(ADP-ribose) polymerase 1(PARP1)이 필요하며, MMEJ의 초기 단계일 수 있습니다. 마이크로호몰로지 영역의 쌍을 이룬 다음, 돌출된 플랩을 제거하기 위한 플랩 구조-특이적 엔도뉴클레아제 1(FEN1)의 모집이 있습니다. 이어서 DNA 말단을 결찰하기 위한 부위에 XRCC1-LIG3를 모집하여 온전한 DNA를 유도합니다. MMEJ는 항상 결손을 동반하므로 MMEJ는 DNA 복구를 위한 돌연변이 유발 경로입니다.[34]
- HR을 사용하려면 동일하거나 거의 동일한 시퀀스가 있어야 중단 복구 템플릿으로 사용할 수 있습니다. 이 복구 과정을 담당하는 효소 기계는 감수분열 동안 염색체 교차를 담당하는 기계와 거의 동일합니다. 이 경로는 자매 염색질(DNA 복제 후 G2에서 이용 가능) 또는 상동 염색체를 주형으로 사용하여 손상된 염색체를 복구할 수 있도록 합니다. 단일 가닥 파손 또는 복구되지 않은 병변에 걸쳐 합성을 시도하는 복제 기계에 의해 발생하는 DSB는 복제 포크의 붕괴를 유발하고 일반적으로 재조합에 의해 복구됩니다.
시험관 내 시스템에서 MMEJ는 HR 및 NHEJ 메커니즘이 모두 사용 가능할 때 HR의 10-20% 수준에서 포유동물 세포에서 발생했습니다.[32]
극호기성 데이노코커스 라디오두란스는 이온화 방사선 및 기타 소스로부터 DNA 손상을 견뎌내는 놀라운 능력을 가지고 있습니다. 무작위로 DNA가 깨지는 유전체의 최소 2개의 사본은 어닐링을 통해 DNA 조각을 형성할 수 있습니다. 부분적으로 겹치는 단편은 상보적인 파트너 가닥이 발견될 때까지 확장을 계속할 수 있는 움직이는 D-루프를 통해 상동 영역의 합성에 사용됩니다. 마지막 단계에서는 RecA 의존적 상동 재조합에 의한 교차가 있습니다.[35]
토포이소머라제는 DNA의 슈퍼코일링 상태를 변화시키는 과정에서 단일 가닥 및 이중 가닥 절단을 모두 도입하며, 이는 특히 개방형 복제 포크 근처의 지역에서 흔히 발생합니다. 토포이소머라제 생화학적 메커니즘에서 자연적인 중간체이고 그것을 만든 효소에 의해 즉시 복구되기 때문에 그러한 분열은 DNA 손상으로 간주되지 않습니다.
DNA 이중 가닥 절단의 또 다른 유형은 DNA 열에 민감하거나 열에 불안정한 부위에서 유래합니다. 이 DNA 부위들은 초기 DSB가 아닙니다. 그러나, 그들은 높은 온도로 처리한 후 DSB로 전환합니다. 이온화 조사는 군집된 손상으로 매우 복잡한 형태의 DNA 손상을 유도할 수 있습니다. DNA 나선의 다양한 위치에 다양한 유형의 DNA 병변으로 구성되어 있습니다. 이러한 밀접하게 위치한 병변 중 일부는 고온에 노출되면 DSB로 전환될 수 있습니다. 하지만 이 병변의 정확한 특성과 상호 작용은 아직 알려지지[36] 않았습니다
트랜스레젼 합성
트랜스 병변 합성(TLS)은 DNA 복제 기계가 티민 이량체 또는 AP 부위와 같은 과거의 DNA 병변을 복제할 수 있도록 하는 DNA 손상 내성 과정입니다.[37] 이것은 특수화된 트랜슬리온 중합효소(즉, Y 중합효소 계열의 DNA 중합효소 IV 또는 V)를 위한 일반적인 DNA 중합효소를 전환하는 것을 포함하며, 종종 손상된 뉴클레오티드 반대쪽 염기의 삽입을 촉진할 수 있는 더 큰 활성 부위를 갖는 것을 포함합니다. 중합효소 전환은 무엇보다도 복제 진행성 인자 PCNA의 번역 후 변형에 의해 매개되는 것으로 생각됩니다. 트랜스 병변 합성 중합효소는 일반 중합효소에 비해 손상되지 않은 주형에 낮은 충실도(잘못된 염기를 삽입하는 경향이 높음)를 갖는 경우가 많습니다. 그러나 많은 사람들이 특정 유형의 손상과 반대되는 정확한 베이스를 삽입하는 데 매우 효율적입니다. 예를 들어, Pol η은 UV 조사에 의해 유도된 병변의 오류 없는 우회를 매개하는 반면, Pol ι은 이러한 부위에 돌연변이를 도입합니다. Pol η은 Watson-Crick 염기쌍을 이용하여 T^T 광이량체를 가로질러 첫 번째 아데닌을 추가하고, 두 번째 아데닌은 Hoogsteen 염기쌍을 이용하여 그 합성 형태로 추가될 것으로 알려져 있습니다. 세포적 관점에서, 전이 합성 동안 점 돌연변이의 도입 위험을 감수하는 것은 총 염색체 수차 또는 세포 사멸을 유발할 수 있는 DNA 복구의 보다 과감한 메커니즘에 의존하는 것이 바람직할 수 있습니다. 간단히 말해서, 이 과정은 DNA 복제가 정지된 위치에서 병변을 우회하거나 복구하는 전문 중합효소를 포함합니다. 예를 들어, 인간 DNA 중합효소 에타는 표적 및 반표적 돌연변이를 유발할 수 있지만 구아닌-티민 가닥 내 가교, G[8,5-Me]T와 같은 복잡한 DNA 병변을 우회할 수 있습니다.[38] Paromita Raychaudhury와 Ashis Basu는[39] 특정 DNA 중합효소 녹아웃으로 대장균에서 G[8,5-Me]T 변형 플라스미드를 복제함으로써 대장균에서 동일한 병변의 독성과 돌연변이 유발을 연구했습니다. 3가지 SOS-유도성 DNA 중합효소인 pol II, pol IV 및 pol V가 결여된 균주에서 생존력이 매우 낮았으며, 이는 트랜스슬리온 합성이 주로 이러한 특수 DNA 중합효소에 의해 수행됨을 나타냅니다. 세포핵항원(PCNA: Prolipating cell nuclear antigen)에 의해 이들 중합효소에 바이패스 플랫폼이 제공됩니다. 정상적인 상황에서는 중합효소에 결합된 PCNA가 DNA를 복제합니다. 병변 부위에서 PCNA는 RAD6/RAD18 단백질에 의해 유비퀴틴화되거나 변형되어 특수 중합효소가 병변을 우회하고 DNA 복제를 재개할 수 있는 플랫폼을 제공합니다.[40][41] 트랜슬리온 합성 후에는 확장이 필요합니다. Pol η의 경우처럼 TLS에 오류가 없는 경우에는 복제 폴리머라제에 의해 이 확장을 수행할 수 있지만, TLS에 오류가 없는 경우에는 이를 확장하기 위해 전문적인 폴리머라제인 Pol ζ가 필요합니다. Pol ζ는 단자의 불일치를 확장할 수 있는 반면에 더 많은 처리 폴리머라제는 확장할 수 없습니다. 따라서 병변이 발생하면 복제 포크가 느려지고, PCNA가 진행성 중합효소에서 Pol ι와 같은 TLS 중합효소로 전환하여 병변을 고정한 다음 PCNA가 Pol ζ로 전환하여 불일치를 연장할 수 있으며, 마지막 PCNA가 진행성 중합효소로 전환하여 복제를 계속합니다.
DNA 손상에 대한 글로벌 대응
이온화 방사선, 자외선 또는 화학 물질에 노출된 세포는 부피가 큰 DNA 병변 및 이중 가닥 파손 부위가 여러 곳에 도달하기 쉽습니다. 더욱이 DNA 손상제는 단백질, 탄수화물, 지질 및 RNA와 같은 다른 생체 분자를 손상시킬 수 있습니다. 구체적으로, 이중 가닥 파손 또는 복제 포크를 지연시키는 부가물의 축적은 DNA 손상에 대한 전 세계적인 반응에 대한 알려진 자극 신호 중 하나입니다.[42] 손상에 대한 전체적인 반응은 세포 자신의 보존을 향한 작용이며, 거대분자 복구, 병변 우회, 내성 또는 세포자멸사의 여러 경로를 유발합니다. 글로벌 반응의 공통적인 특징은 다중 유전자의 유도, 세포 주기 정지, 세포 분열 억제입니다.
초기단계
진핵생물 DNA를 염색질로 포장하는 것은 효소를 작용 부위로 모집해야 하는 모든 DNA 기반 과정에 장벽을 제공합니다. DNA 복구를 허용하려면 염색질을 리모델링해야 합니다. 진핵생물에서 ATP 의존적 염색질 리모델링 복합체와 히스톤 변형 효소는 이 리모델링 과정을 수행하는 데 사용되는 주요 요소입니다.[43]
염색질 이완은 DNA 손상 부위에서 빠르게 발생합니다.[44][45] 스트레스 활성화 단백질 키나제인 c-Jun N-말단 키나제(JNK)는 이중 가닥 파손 또는 기타 DNA 손상에 대한 반응으로 세린 10 상의 SIRT6을 인산화합니다.[46] 이러한 번역 후 변형은 SIRT6를 DNA 손상 부위로 동원하는 것을 용이하게 하며, 폴리(ADP-리보스) 중합효소1(PARP1)을 DNA 파손 부위로 효율적으로 모집하고 DSB를 효율적으로 복구하는 데 필요합니다.[46] PARP1 단백질은 DNA 손상 부위에 1초 이내에 나타나기 시작하며, 손상이 발생한 후 1.6초 이내에 절반의 최대 축적량을 기록합니다.[47] PARP1은 자체적으로 고분자 아데노신 이인산 리보스(폴리(ADP-리보스) 또는 PAR) 사슬을 합성합니다. 다음으로 염색질 리모델링기 ALC1은 폴리-ADP 리보오스 사슬인 PARP1 작용 생성물에 빠르게 부착되고 ALC1은 손상 발생 후 10초 이내에 DNA 손상에 도달을 완료합니다.[45] ALC1의 작용으로 추정되는 최대 염색질 이완의 약 절반이 10초에 발생합니다.[45] 이를 통해 DNA 복구 효소 MRE11을 모집하여 13초 이내에 DNA 복구를 시작할 수 있습니다.[47]
γH2AX의 인산화된 형태인 H2AX도 DNA 이중 가닥이 끊어지면 염색질이 축합되는 초기 단계에 관여합니다. 히스톤 변이 H2AX는 인간 염색질에서 H2A 히스톤의 약 10%를 구성합니다.[48] γH2AX(세린 139에 인산화된 H2AX)는 세포의 조사 후 20초 이내에 (DNA 이중 가닥 분열 형성으로) 검출될 수 있으며, γH2AX의 반치 최대 축적은 1분 안에 발생합니다. 인산화된 γH2AX를 가진 염색질의 범위는 DNA 이중 가닥 절단 부위에서 약 200만 염기쌍입니다. γH2AX는 그 자체로 염색질 탈응축을 일으키지 않지만, 조사 후 30초 이내에 RNF8 단백질이 γH2AX와 연관되어 검출될 수 있습니다. RNF8은 뉴클레오솜 리모델링 및 탈아세틸화효소 복합체 NuRD의 구성요소인 [50]CHD4와의 후속 상호작용을 통해 광범위한 염색질 탈축합을 매개합니다.
DDB2는 DDB1과 이종이량체 복합체에서 발생합니다. 이 복합체는 유비퀴틴 연결효소 단백질 CUL4A[51] 및 PARP1과 추가로 복합체를 형성합니다.[52] 이 더 큰 복합체는 염색질 내에서 자외선으로 인한 손상과 빠르게 연관되며, 반 최대 연관성은 40초 만에 완료됩니다.[51] DDB1과 DDB2에 모두 부착된 PARP1 단백질은 DDB2에 DNA 리모델링 단백질 ALC1을 끌어당기는 폴리-ADP 리보스 사슬을 형성합니다.[52] ALC1의 작용은 DNA의 UV 손상 부위의 염색질을 이완시킵니다. 이 이완은 뉴클레오티드 절제 복구 경로의 다른 단백질이 염색질로 들어가 UV 유도 시클로부탄 피리미딘 이량체 손상을 복구할 수 있게 합니다.
신속한 염색질 리모델링 후 세포 주기 체크포인트가 활성화되어 세포 주기가 진행되기 전에 DNA 복구가 이루어질 수 있습니다. 먼저 DNA가 손상된 후 5분 또는 6분 이내에 ATM과 ATR 두 가지 키나제가 활성화됩니다. 이어서 세포주기 체크포인트 단백질 Chk1의 인산화가 진행되어 DNA가 손상된 후 약 10분 후에 그 기능이 시작됩니다.[53]
DNA 손상 검사점
DNA 손상 후 세포 주기 체크포인트가 활성화됩니다. 체크포인트 활성화는 세포 주기를 일시 중지시키고 세포가 분열을 계속하기 전에 손상을 복구할 수 있는 시간을 줍니다. DNA 손상 검사점은 G1/S 및 G2/M 경계에서 발생합니다. S내 체크포인트도 존재합니다. 체크포인트 활성화는 ATM과 ATR의 두 마스터 키나제에 의해 제어됩니다. ATM은 DNA 이중 가닥 파손과 염색질 구조의 [54]파괴에 반응하는 반면 ATR은 주로 정체된 복제 포크에 반응합니다. 이러한 키나제는 신호 전달 캐스케이드에서 다운스트림 표적을 인산화하여 결국 세포 주기 정지로 이어집니다. BRCA1, MDC1 및 53BP1을 포함하는 체크포인트 매개 단백질 클래스도 확인되었습니다.[55] 이러한 단백질은 체크포인트 활성화 신호를 다운스트림 단백질로 전달하는 데 필요한 것으로 보입니다.
DNA 손상 체크포인트는 G1, G2, 메타기의 세포 주기 진행을 차단하고 DNA가 손상되면 S상 진행 속도를 늦추는 신호전달 경로입니다. 세포 주기의 일시 정지로 이어져 세포가 분열하기 전에 손상을 복구할 수 있습니다.
체크포인트 단백질은 포스파티딜이노시톨 3-키나제(PI3K) 유사 단백질 키나제, 증식 세포 핵항원(PCNA) 유사 그룹, 2개의 세린/트레오닌(S/T) 키나제 및 이들의 어댑터의 네 그룹으로 분리될 수 있습니다. 모든 DNA 손상 유도 체크포인트 반응의 중심은 진화 과정에서 서열과 기능이 잘 보존된 첫 번째 그룹인 PI3K 유사 단백질 키나제-ATM(Ataxia telangiectasia 돌연변이) 및 ATR(Ataxia 및 Rad 관련) 키나제에 속하는 한 쌍의 큰 단백질 키나제입니다. 모든 DNA 손상 반응은 DNA 손상 부위의 염색체와 결합할 수 있고, DNA 손상 반응 성분과 DNA 복구 복합체가 조립될 수 있는 플랫폼인 부속 단백질과 결합할 수 있기 때문에 ATM이나 ATR이 필요합니다.
ATM과 ATR의 중요한 다운스트림 표적은 p53인데, 이는 DNA 손상에 따른 세포자멸사 유도에 필요하기 때문입니다.[56] 사이클린 의존성 키나제 억제제 p21은 p53 의존성 및 p53 의존성 메커니즘에 의해 유도되며, 사이클린/사이클린 의존성 키나제 복합체를 비활성화함으로써 G1/S 및 G2/M 체크포인트에서 세포 주기를 정지시킬 수 있습니다.[57]
원핵생물 SOS 반응은
SOS 반응은 광범위한 DNA 손상에 대한 반응으로 대장균과 다른 박테리아의 유전자 발현 변화입니다. 원핵생물 SOS 시스템은 LexA와 RecA의 두 가지 핵심 단백질에 의해 조절됩니다. LexA 동형이량체는 일반적으로 SOS 상자라고 하는 오퍼레이터 서열에 결합하는 전사 억제인자입니다. 대장균에서 LexA는 lexA와 recA 유전자를 포함한 약 48개 유전자의 전사를 조절하는 것으로 알려져 있습니다.[58] SOS 반응은 박테리아 영역에 널리 퍼져 있는 것으로 알려져 있지만 Spirochetes와 같은 일부 박테리아 문에는 대부분 존재하지 않습니다.[59] SOS 반응을 활성화하는 가장 일반적인 세포 신호는 단일 가닥 DNA(ssDNA) 영역으로, 정체된 복제 포크 또는 이중 가닥 파손으로 인해 발생하며, 이들은 DNA 헬리카제에 의해 처리되어 두 DNA 가닥을 분리합니다.[42] 개시 단계에서 RecA 단백질은 ATP 가수분해 유도 반응에서 ssDNA에 결합하여 RecA-ssDNA 필라멘트를 생성합니다. RecA-ssDNA 필라멘트는 LexA 자가단백질분해효소 활성을 활성화하여 궁극적으로 LexA 이량체의 절단 및 후속 LexA 분해로 이어집니다. LexA 억제인자의 손실은 SOS 유전자의 전사를 유도하고 추가 신호 유도, 세포 분열 억제 및 손상 처리를 담당하는 단백질 수준의 증가를 허용합니다.
대장균에서 SOS 상자는 회문 구조와 높은 서열 보존도를 가진 프로모터 근처의 20개 뉴클레오티드 긴 서열입니다. 다른 부류와 문에서 SOS 상자의 서열은 길이와 구성에 따라 상당히 다르지만 항상 고도로 보존되어 있으며 게놈에서 가장 강한 짧은 신호 중 하나입니다.[59] SOS 상자의 높은 정보 함량은 LexA가 다른 프로모터에 차등적으로 결합할 수 있도록 하고 SOS 반응의 타이밍을 허용합니다. 병변 복구 유전자는 SOS 반응 초기에 유도됩니다. UmuCD'2(DNA 중합효소 V라고도 함)와 같은 오류가 발생하기 쉬운 트랜스 병변 중합효소는 나중에 최후의 수단으로 유도됩니다.[60] 중합효소를 이용하거나 재조합을 통해 DNA 손상이 복구되거나 우회되면 세포 내 단일가닥 DNA의 양이 감소하여 RecA 필라멘트의 양을 낮추면 LexA 동형이량체의 절단 활성이 감소하고, 이는 프로모터 근처의 SOS 상자에 결합하여 정상적인 유전자 발현을 회복합니다.
DNA 손상에 대한 진핵생물 전사 반응
DNA 손상제에 노출된 진핵 세포는 또한 DNA 복구, 세포 주기 체크포인트 제어, 단백질 밀매 및 분해에 관여하는 여러 단백질을 유도하여 중요한 방어 경로를 활성화합니다. 이러한 게놈 전체의 전사 반응은 매우 복잡하고 엄격하게 조절되므로 손상에 대한 조정된 글로벌 반응이 가능합니다. 효모 Saccharomyces cerevisiae를 DNA 손상제에 노출시키면 전사 프로파일이 겹치지만 구별됩니다. 환경적 충격반응과 유사성은 전사 활성화 수준에서 일반적인 글로벌 스트레스 반응 경로가 존재함을 나타냅니다. 대조적으로, 다른 인간 세포 유형은 손상에 대해 다르게 반응하며, 이는 공통적인 세계적인 반응이 없음을 나타냅니다. 효모와 인간 세포의 이러한 차이에 대한 가능한 설명은 포유류 세포의 이질성에 있을 수 있습니다. 동물에서는 DNA 손상에 대한 민감도가 서로 다르게 진화한 여러 기관들 사이에 여러 종류의 세포들이 분포되어 있습니다.[61]
일반적으로 DNA 손상에 대한 글로벌 반응은 복제 후 복구, 상동 재조합, 뉴클레오티드 절제 복구, DNA 손상 체크포인트, 글로벌 전사 활성화, mRNA 붕괴를 제어하는 유전자 등을 담당하는 여러 유전자의 발현을 포함합니다. 세포가 대량으로 손상되면 세포사멸을 겪고 죽거나, 변형된 유전체를 가지고 생활하는 대가로 살아남는 것과 같은 중요한 결정을 남깁니다. 손상에 대한 내성이 증가하면 생존율이 증가하여 돌연변이가 더 많이 축적될 수 있습니다. 효모 Rev1 및 인간 중합효소 η는 DNA 손상에 대한 글로벌 반응 동안 존재하는 Y 패밀리 트랜스레젼 DNA 중합효소의 구성체이며 진핵생물의 DNA 손상에 대한 글로벌 반응 동안 향상된 돌연변이 유발을 담당합니다.
노화
DNA 복구 불량으로 인한 병리학적 영향

DNA 복구에 유전적 결함이 있는 실험동물은 수명이 줄어들고 암 발생률이 증가하는 경우가 많습니다.[13] 예를 들어, 지배적인 NHEJ 경로와 텔로미어 유지 메커니즘이 결핍된 쥐는 림프종과 감염이 더 자주 발생하고, 그 결과 야생형 쥐보다 수명이 짧습니다.[62] 마찬가지로 DNA 나선을 풀어주는 핵심 복구 및 전사 단백질이 결핍된 쥐는 노화 관련 질병의 조기 발병과 그에 따른 수명 단축을 겪습니다.[63] 그러나, 모든 DNA 복구 결핍이 정확히 예측된 효과를 만들어내는 것은 아닙니다; NER 경로가 결핍된 쥐는 그에 상응하는 더 높은 돌연변이 비율 없이 단축된 수명을 나타냈습니다.[64]
쥐, 벌거벗은 두더지쥐, 인간의 최대 수명은 각각 ~3년, ~30년, ~129년입니다.[65] 이 중 가장 짧은 수명을 가진 종인 쥐는 여러 DNA 복구 경로의 핵심 유전자를 포함한 DNA 복구 유전자를 인간과 벌거벗은 두더지 쥐보다 낮은 수준으로 발현합니다.[65] 또한 인간과 벌거벗은 두더지 쥐의 여러 DNA 복구 경로는 쥐에 비해 상향 조절됩니다. 이러한 관찰은 DNA 복구가 증가하면 수명이 연장된다는 것을 시사합니다.[65]
DNA 손상 속도가 세포가 복구할 수 있는 능력을 넘어서면 오류의 축적이 세포를 압도해 조기노화나 세포사멸, 암을 유발할 수 있습니다. DNA 복구 기능의 결함과 관련된 유전 질환은 조기 노화,[13] 발암 물질에 대한 민감도 증가 및 그에 따른 암 위험 증가를 초래합니다(아래 참조). 반면에, 가장 방사선에 강한 유기체인 Deinococcus radiodurans와 같이 DNA 복구 시스템이 강화된 유기체는 DNA 복구의 효율성과 특히 NHEJ의 향상으로 인해 방사능의 이중 가닥 파괴 유도 효과에 대해 현저한 내성을 나타냅니다.[66]
장수 및 칼로리 제한

많은 개별 유전자들이 유기체 집단 내에서 수명의 변화에 영향을 미치는 것으로 확인되었습니다. 이러한 유전자의 영향은 환경, 특히 유기체의 식단에 크게 의존합니다. 칼로리 제한은 아마도 영양소 감지 경로와 대사 속도 감소를 통해 다양한 유기체에서 재현 가능하게 수명 연장을 초래합니다. 이러한 제한이 수명 연장을 초래하는 분자 메커니즘은 아직 명확하지 않습니다(일부 논의는 참조[67]). 그러나 DNA 복구에 관여하는 것으로 알려진 많은 유전자의 행동은 칼로리 제한 조건에서 변경됩니다. 항노화 특성을 갖는 것으로 보고된 여러 제제는 대사 활성의 감소의 증거인 mTOR 신호 전달의 구성 수준을 약화시키고 동시에 내인성 생성 활성 산소 종에 의해 유도된 DNA 손상의 구성 수준을 감소시키는 것으로 나타났습니다.[68]
예를 들어 선충인 예쁜꼬마선충에서 DNA 포장을 조절하는 유전자 SIR-2의 유전자 투여량을 늘리면 수명이 크게 연장될 수 있습니다.[69] SIR-2의 포유류 상동체는 NHEJ에 관여하는 다운스트림 DNA 복구 인자를 유도하는 것으로 알려져 있으며, 이는 칼로리 제한 조건에서 특히 촉진되는 활성입니다.[70] 열량 제한은 설치류의 핵 DNA에서 염기 절제 복구 속도와 밀접한 관련이 있지만 미토콘드리아 DNA에서는 유사한 효과가 관찰되지 않았습니다.[71][72]
DNA 복구 경로의 업스트림 이펙터인 C. elegans 유전자 AGE-1은 자유 섭식 조건에서 극적으로 연장된 수명을 부여하지만 칼로리 제한 조건에서는 생식 적합성을 감소시킵니다.[73] 이 관찰은 노화의 생물학적 기원에 대한 다면발현 이론을 뒷받침하는데, 이는 생애 초기에 큰 생존 이점을 부여하는 유전자는 말년에 상응하는 단점을 지니고 있더라도 선택될 것임을 시사합니다.
의약품 및 DNA 복구 변조
유전성 DNA 복구 장애
NER 메커니즘의 결함은 다음과 같은 몇 가지 유전적 장애의 원인이 됩니다.
- 색소성 피부염: 햇빛/자외선에 과민하여 피부암 발생 증가 및 조기노화를 초래합니다.
- 코케인 증후군 : 자외선과 화학작용제에 대한 과민반응
- 트리코티오디스트로피: 민감한 피부, 부서지기 쉬운 모발 및 손톱
정신 지체는 종종 후자의 두 가지 장애를 동반하여 발달 뉴런의 취약성이 증가함을 시사합니다.
다른 DNA 복구 장애는 다음과 같습니다.
- 베르너 증후군: 조기 노화와 성장 지연
- 블룸스 증후군: 햇빛 과민성, 높은 악성 종양(특히 백혈병) 발생률.
- 전리방사선 및 일부 화학작용제에 대한 운동실조증(Ataxia telangiectasia): 전리방사선 및 일부 화학작용제에 대한 민감성
위의 질병들은 모두 '분절성 노화증'(segmental progeria, 가속노화질환)이라고 불리는데, 이는 영향을 받는 사람들이 고령으로 나타나고 비정상적으로 어린 나이에 노화와 관련된 질병을 경험하는 반면, 노쇠의 모든 증상을 나타내지 않기 때문입니다.
DNA 복구 기능 저하와 관련된 다른 질병으로는 판코니 빈혈, 유전성 유방암, 유전성 대장암 등이 있습니다.
암
DNA 복구 메커니즘에 내재된 한계 때문에 인간이 충분히 오래 살았다면 결국 모두 암에 걸릴 것입니다.[74][75] 암 위험을 높이는 유전자 복구 유전자 돌연변이는 최소 34개입니다. 이러한 돌연변이들 중 많은 부분이 DNA 복구를 정상보다 덜 효과적으로 만듭니다. 특히 유전성 비다형성 대장암(HNPCC)은 DNA 불일치 복구 경로의 특정 돌연변이와 강하게 연관되어 있습니다. 돌연변이가 보균자에게 유방암 위험을 크게 증가시키는 두 가지 중요한 유전자인 BRCA1과 BRCA2는 [76]모두 많은 수의 DNA 복구 경로, 특히 NHEJ 및 상동 재조합과 관련이 있습니다.
항암치료, 방사선치료 등 암치료 시술은 세포가 DNA 손상을 복구하는 능력을 압도해 세포가 사망하는 결과를 낳습니다. 가장 빠르게 분열하고 있는 세포들 – 가장 일반적으로 암세포들 – 가 우선적으로 영향을 받습니다. 부작용은 장, 피부, 조혈계에 있는 전구세포와 같이 암은 아니지만 빠르게 분열하는 다른 세포들도 영향을 받는다는 것입니다. 현대 암 치료법은 물리적인 방법(종양 부위에 치료제를 집중시키는 것) 또는 생화학적인 방법(몸의 암세포에 고유한 특징을 이용하는 것)을 통해 암과 관련된 세포와 조직의 DNA 손상을 국소화하려고 시도합니다. DNA 손상 반응 유전자를 표적으로 하는 치료법의 맥락에서 후자의 접근법은 '합성 치사율'이라고 불립니다.[77]
아마도 이러한 '합성 치사율' 약물 중 가장 잘 알려진 것은 폴리(ADP-ribose) 중합효소 1(PARP1) 억제제 올라파립(olaparib)으로, 2015년 식품의약국으로부터 BRCA 결핍 난소암 여성의 치료를 승인받았습니다. DNA 손상 반응(특히 상동 재조합 복구)이 부분적으로 손실된 종양 세포는 부분적으로 PARP1 유전자 산물로 구성된 메커니즘인 단일 가닥 파손 복구(single strand break repair)라는 또 다른 메커니즘에 의존합니다.[78] 올라파립은 화학 요법과 결합하여 병용 화학 요법으로 인한 DNA 손상으로 인한 단일 가닥 파손 복구를 억제합니다. 이 잔류 DNA 복구 메커니즘에 의존하는 종양 세포는 손상을 복구할 수 없기 때문에 생존 및 증식할 수 없는 반면, 정상 세포는 기능하는 상동 재조합 메커니즘으로 손상을 복구할 수 있습니다.
암에서 흔히 발견되는 다른 잔류 DNA 복구 메커니즘에 사용하기 위한 많은 다른 약물들이 현재 조사 중에 있습니다. 그러나 DNA 손상 반응 경로의 재배선 및 이전에 억제된 결함의 복귀를 통해 달성된 획득 내성의 새로운 증거로 인해 합성 치사율 치료 접근법에 의문이 제기되었습니다.[79]
암의 DNA 복구 결함
DNA 손상 반응이 재생 전 세포의 악성 변형에 대한 장벽으로 작용한다는 것은 지난 몇 년 동안 명백해졌습니다.[80] 이전 연구에서는 종양 유전자 활성화[81] 및 재생 전 결장 선종이 있는 세포 배양 모델에서 DNA 손상 반응이 증가했음을 보여주었습니다.[82] DNA 손상 반응 메커니즘은 세포 주기 정지를 유발하고, 복구가 불가능한 경우 DNA 병변을 복구하거나 세포 사멸/노화를 촉진하려고 시도합니다. 종양성 돌연변이로부터 증가된 증식 신호로 인해 재생 스트레스가 재생 전 세포에서 관찰됩니다. 복제 스트레스는 복제 개시/원점 발화 증가, 전사-복제 복합체의 전사 및 충돌 증가, 뉴클레오티드 결핍, 활성 산소종(ROS) 증가를 특징으로 합니다.[83]
복제 스트레스는 종양의 진화에서 DNA 손상 반응 유전자의 돌연변이를 비활성화하기 위한 선택과 [84]함께 일부 DNA 손상 반응 메커니즘의 하향 조절 및/또는 손실로 이어져 DNA 복구 및/또는 노화/프로그램된 세포 사망을 초래합니다. 실험용 쥐 모델에서 DNA 손상 반응 매개 세포 노쇠의 손실은 짧은 헤어핀 RNA(shRNA)를 사용하여 이중 가닥 절단 반응 키나아제(ataxia telangiectasia, ATM)를 억제한 후 관찰되어 종양 크기와 침습성이 증가했습니다.[82] 유전적으로 DNA 복구 메커니즘에 결함을 가지고 태어난 인간(예를 들어, Li-Fraumeni 증후군)은 암 위험이 더 높습니다.[85]
DNA 손상 반응 돌연변이의 유병률은 암 종류에 따라 다릅니다. 예를 들어, 유방 침습성 암종의 30%는 상동 재조합에 관여하는 유전자에 돌연변이가 있습니다.[80] 암에서 하향 조절은 모든 DNA 손상 반응 메커니즘(염기 절제 복구(BER), 뉴클레오티드 절제 복구(NER), DNA 불일치 복구(MMR), 상동 재조합 복구(HR), 비상동 말단 접합(NHEJ) 및 전사 DNA 합성(TLS)에 걸쳐 관찰됩니다.[86] DNA 손상 복구 유전자에 대한 돌연변이뿐만 아니라 DNA 손상 복구가 일어날 수 있는 충분한 시간을 허용하기 위해 세포 주기를 억제하는 역할을 하는 유전자에도 돌연변이가 발생하고, 일부 유전자는 DNA 손상 복구와 세포 주기 체크포인트 제어에 모두 관여하고, 예를 들어 ATM 및 체크포인트 키나제 2(CHEK2) – 비소세포 폐암에서 종종 없거나 하향 조절되는 종양 억제제.[87]
| 인사 | NHEJ | SSA | FA | BER | NER | MMR | |
|---|---|---|---|---|---|---|---|
| 현금 자동 입출금기 | |||||||
| ATR | |||||||
| 팍스IP | |||||||
| RPA | |||||||
| BRCA1 | |||||||
| BRCA2 | |||||||
| RAD51 | |||||||
| RFC | |||||||
| XRCC1 | |||||||
| PCNA | |||||||
| PARP1 | |||||||
| ERCC1 | |||||||
| MSH3 |
암의 후성유전학적 DNA 복구 결함
고전적으로, 암은 종양 억제 유전자와 종양 유전자의 돌연변이, 염색체 이상을 포함하는 진행성 유전적 이상에 의해 유발되는 일련의 질병으로 간주되어 왔습니다. 그러나 암도 후성유전학적 변화에 의해 발생한다는 사실이 밝혀졌습니다.[88]
후성유전학적 변화는 뉴클레오티드 서열의 변화를 수반하지 않는 유전체에 대한 기능적으로 관련된 변형을 말합니다. 이러한 변형의 예로는 DNA 메틸화(hypermethylation 및 hypomethylation) 및 히스톤 변형의 변화, 염색체 구조의 변화([89]HMGA2 또는 HMGA1과 같은 단백질의 부적절한 발현에 의해 야기됨)[90] 및 마이크로RNA에 의한 변화가 있습니다. 이러한 후성유전학적 변화는 기본 DNA 서열을 변경하지 않고 유전자 발현을 조절하는 역할을 합니다. 이러한 변화는 일반적으로 세포 분열을 통해 유지되며, 여러 세포 세대 동안 지속되며, 후성 돌연변이(돌연변이에 해당함)로 간주될 수 있습니다.
암에서 후성유전학적 변화가 많이 발견되지만 DNA 복구 단백질의 발현 감소를 유발하는 DNA 복구 유전자의 후성유전학적 변화가 특히 중요한 것으로 보입니다. 이러한 변화는 암으로 진행하는 초기에 발생하는 것으로 생각되며, 암의 유전적 불안정성의 원인이 될 가능성이 있습니다.[91][92][93]
DNA 복구 유전자의 발현 감소는 부족한 DNA 복구를 야기합니다. DNA 복구가 결핍되면 DNA 손상은 평상시보다 높은 수준으로 세포 내에 남아 있으며 이러한 과도한 손상은 돌연변이나 후성 돌연변이의 빈도를 증가시킵니다. 돌연변이율은 DNA 불일치 복구[94][95] 또는 상동 재조합 복구(HRR)에서 결함이 있는 세포에서 상당히 증가합니다.[96] HRR 결함 세포에서도 염색체 재배열과 이수체가 증가합니다.[97]
DNA 손상 수준이 높으면 돌연변이가 증가할 뿐만 아니라 발적도 증가합니다. DNA 이중 가닥이 끊어지거나 다른 DNA 손상이 복구되는 동안 복구 부위가 불완전하게 제거되면 후생유전자 침묵이 발생할 수 있습니다.[98][99]
유전자 돌연변이로 DNA 복구 단백질 발현이 부족하면 암 발생 위험이 높아질 수 있습니다. 34개의 DNA 복구 유전자(DNA 복구-결핍 장애 참조) 중 하나에 유전적 장애가 있는 사람은 암 위험이 증가하며, 일부 결함은 평생 암 확률을 최대 100%(예: p53 돌연변이)까지 유발합니다.[100] 하지만, 이러한 생식선 돌연변이(침투성 암 증후군을 유발하는)는 암의 약 1%만 발생시키는 원인입니다.[101]
DNA 복구 유전자의 에피뮤테이션 빈도

DNA 복구 효소의 결핍은 DNA 복구 유전자에서 새로 발생하는 체세포 돌연변이에 의해 발생하는 경우도 있지만, DNA 복구 유전자의 발현을 감소시키거나 침묵시키는 후생유전적 변화에 의해 발생하는 경우가 훨씬 더 많습니다. 예를 들어, 113개의 대장암을 순차적으로 검사했을 때, DNA 복구 유전자 MGMT에서 4개만이 미스센스 돌연변이를 가졌고, 대다수는 MGMT 프로모터 영역의 메틸화(후성유전학적 변형)로 인해 MGMT 발현이 감소했습니다.[102] 5개의 다른 연구에서 대장암의 40%에서 90% 사이에서 MGMT 촉진체 영역의 메틸화로 인해 MGMT 발현이 감소하는 것으로 나타났습니다.[103][104][105][106][107]
마찬가지로 DNA 복구 유전자 PMS2 발현이 결여된 불일치 복구결핍 대장암 119건 중 PMS2 유전자의 돌연변이로 인해 6건에서 PMS2가 결핍되었고, 103 사례에서 PMS2 발현은 프로모터 메틸화로 인해 페어링 파트너 MLH1이 억제되었기 때문에 결함이 있었습니다(PMS2 단백질은 MLH1이 없을 때 불안정함).[108] 다른 10개의 경우에서 PMS2 발현의 손실은 MLH1을 하향 조절하는 microRNA인 miR-155의 후성유전학적 과발현으로 인한 것일 수 있습니다.[109]
추가적인 예로, 후성유전학적 결함이 다양한 암(예: 유방, 난소, 대장 및 두경부)에서 발견되었습니다. Facista 등에 의해 평가된 49개의 대장암 중 대다수에서 ERCC1, XPF 또는 PMS2의 발현에 있어서 2개 또는 3개의 결함이 동시에 발생합니다.[110]
이 섹션의 차트는 빈번한 DNA 손상제, DNA 병변의 예 및 이러한 DNA 손상을 처리하는 경로를 보여줍니다. 적어도 169개의 효소가 DNA 복구에 직접 사용되거나 DNA 복구 과정에 영향을 미칩니다.[111] 이 중 83개는 차트에 표시된 5종의 DNA 손상을 복구하는 데 직접 사용됩니다.[citation needed]
이러한 복구 과정의 중심이 되는 더 잘 연구된 유전자 중 일부가 도표에 나와 있습니다. 적색, 회색 또는 청록색으로 표시된 유전자 지정은 다양한 유형의 암에서 후성유전적으로 자주 변경되는 유전자를 나타냅니다. 빨간색, 회색 또는 청록색으로 강조된 각각의 유전자에 대한 위키피디아 기사는 후생유전적 변화와 이러한 후생이 발견되는 암에 대해 설명합니다. 리뷰 [112]기사와 광범위한 실험 조사 기사는[113][114] 또한 암에서 이러한 후성유전학적 DNA 복구 결함의 대부분을 기록합니다.
다양한 암에서 후성유전학적 메커니즘에 의해 적색으로 강조된 유전자가 감소하거나 침묵하는 경우가 많습니다. 이 유전자들이 발현량이 적거나 없을 때 DNA 손상이 누적될 수 있습니다. 이러한 손상(트랜스레젼 합성 참조)을 지난 복제 오류는 돌연변이를 증가시키고 궁극적으로 암을 유발할 수 있습니다. 정확한 DNA 복구 경로에서 DNA 복구 유전자의 후생유전적 억제는 발암의 중심인 것으로 보입니다.
회색으로 강조된 두 유전자 RAD51 및 BRCA2는 상동 재조합 복구에 필요합니다. 어떤 암에서는 후성유전적으로 과발현되기도 하고 어떤 암에서는 과소발현되기도 합니다. RAD51 및 BRCA2에 대한 위키피디아 기사에서 알 수 있듯이, 이러한 암은 일반적으로 다른 DNA 복구 유전자에 후생유전적 결함이 있습니다. 이러한 복구 결함은 복구되지 않은 DNA 손상을 증가시킬 가능성이 있습니다. 이러한 암에서 볼 수 있는 RAD51 및 BRCA2의 과발현은 보상적 RAD51 또는 BRCA2 과발현에 대한 선택적 압력을 반영하고 상동 재조합 복구를 증가시켜 이러한 과발현 DNA 손상을 적어도 부분적으로 처리할 수 있습니다. RAD51 또는 BRCA2가 과소발현된 경우, 이는 그 자체로 복구되지 않은 DNA 손상을 증가시킵니다. 이러한 손상(트랜스레전 합성 참조)을 지난 복제 오류는 돌연변이와 암을 증가시킬 수 있으므로 RAD51 또는 BRCA2의 과소발현은 그 자체로 발암성이 있습니다.
청록색으로 강조된 유전자는 미세 상동성 매개 말단 결합(MMEJ) 경로에 있으며 암에서 상향 조절됩니다. MMEJ는 이중 가닥 파손에 대한 오류가 발생하기 쉬운 추가 복구 경로입니다. 이중 가닥 파손의 MMEJ 복구에서 쌍을 이루는 양쪽 가닥 사이에 5-25개의 상보적 염기쌍의 상동성은 가닥을 정렬하는 데 충분하지만 일치하지 않는 말단(플랩)이 일반적으로 존재합니다. MMEJ는 가닥이 연결된 여분의 뉴클레오티드(플랩)를 제거한 다음 가닥을 연결하여 온전한 DNA 이중나선을 만듭니다. MMEJ는 거의 항상 최소한 작은 삭제를 포함하므로 돌연변이 유발 경로입니다.[24] MMEJ의 플랩 엔도뉴클레아제인 FEN1은 촉진체 저메틸화에 의해 후생적으로 증가하며 유방, [115]전립선,[116] 위,[117][118] 신경아세포종,[119] 췌장 [120]및 폐의 대부분의 암에서 과발현됩니다.[121] PARP1은 또한 프로모터 영역 ETS 부위가 후성적으로 저메틸화될 때 과발현되며, 이는 자궁내막암[122] 및 BRCA 돌연변이 장성 난소암으로의 진행에 기여합니다.[123] MMEJ 경로의 다른 유전자들 또한 다수의 암에서 과발현되며(요약은 MMEJ 참조), 시안에도 표시됩니다.
인간 체세포에서 DNA 복구의 유전체 전체 분포
인간 게놈의 다양한 영역에 걸친 DNA 복구 경로의 차등 활동은 돌연변이가 종양 게놈 내에 매우 불균일하게 분포되도록 합니다.[124][125] 특히, 인간 게놈의 유전자가 풍부하고 초기에 복제되는 영역은 유전자가 부족하고 후기에 복제되는 헤테로크로마틴보다 낮은 돌연변이 빈도를 보입니다. 이를 뒷받침하는 한 가지 메커니즘은 히스톤 변형 H3K36me3를 포함하며,[126] 이는 불일치 복구 단백질을 모집하여 H3K36me3 표시 영역에서 돌연변이율을 낮출 수 있습니다.[127] 또 다른 중요한 메커니즘은 뉴클레오티드 절제 복구에 관한 것으로, 이는 전사 기계에 의해 모집될 수 있으며, 활성 유전자[125] 및 기타 개방 염색질 영역에서 체세포 돌연변이율을 낮춥니다.[128]
DNA 복구로 인한 후성유전학적 변화
DNA의 손상은 매우 일반적이며 지속적으로 수리되고 있습니다. 후성유전학적 변화는 산화 손상의 DNA 복구 또는 이중 가닥 파손을 동반할 수 있습니다. 인간 세포에서, 산화적 DNA 손상은 하루에 약 10,000번 발생하고, DNA 이중 가닥 파손은 체세포 복제 세포에서 세포 주기에 약 10-50번 발생합니다(DNA 손상(자연적으로 발생함) 참조). DNA 복구의 선택적인 이점은 세포가 DNA 손상에 직면하여 생존할 수 있도록 하는 것입니다. DNA 복구와 함께 발생하는 후성유전학적 변화의 선택적 이점은 명확하지 않습니다.[citation needed]
산화적 DNA 손상을 복구하면 후성유전학적 표지가 변경될 수 있습니다.
정상 상태(내인성 손상이 발생하여 복구됨)에서 평균적인 포유동물 세포 DNA에는 8-옥소-2'-데옥시구아노신(8-OHdG)을 형성하는 약 2,400개의 산화적으로 손상된 구아닌이 있습니다.[129] 8-OHdG는 DNA에 일반적으로 존재하는 산화적 손상의 약 5%를 구성합니다.[130] 산화된 구아닌은 DNA의 모든 구아닌 중에서 무작위로 발생하지 않습니다. 메틸화된 CpG 부위(사이토신이 뒤따르고 5' → 3' 방향을 따라 구아닌이 메틸화되는 곳)에서 구아닌에 대한 서열 선호도가 있습니다(5-mCpG). 5-mCpG 부위는 구아닌 산화에 대한 이온화 전위가 가장 낮습니다.

산화된 구아닌은 오쌍성 잠재력을 가지고 있으며 돌연변이를 유발합니다.[133] OGG1(Oxoguanine glycosylase)은 DNA 복구 동안 산화된 구아닌을 절제하는 역할을 하는 주요 효소입니다. OGG1은 몇 초 안에 8-OHdG를 찾아 결합합니다.[134] 그러나 OGG1은 8-OHdG를 즉시 제거하지 않습니다. HeLa 세포에서 8-OHdG의 최대 절반 제거는 30분 안에 일어나며,[135] 조사된 쥐에서는 쥐 간에서 유도된 8-OHdG가 11분의 반감기로 제거됩니다.[130]
OGG1이 메틸화된 CpG 부위 내에서 산화된 구아닌에 존재하면 TET1을 8-OHdG 병변으로 모집합니다(그림 참조). 이를 통해 TET1은 인접한 메틸화된 시토신을 탈메틸화할 수 있습니다. 시토신의 탈메틸화는 후생유전학적 변화입니다.[citation needed]
예를 들어, 인간 유선 상피세포를22 6시간 동안 HO로 처리했을 때, 8-OHdG는 DNA에서 약 3.5배 증가했고, 이로 인해 유전체 내 5-메틸사이토신의 약 80%가 탈메틸화되었습니다.[132] TET 효소 활성에 의한 유전자 프로모터의 CpGs의 탈메틸화는 메신저 RNA로의 유전자 전사를 증가시킵니다.[136] HO로22 처리된 세포에서, 하나의 특정한 유전자인 BACE1을 조사했습니다.[132] BACE1 CpG 섬의 메틸화 수준은 감소되었고(후성유전학적 변화) 이것은 BACE1 전령 RNA의 발현을 약 6.5배 증가시켰습니다.[citation needed]
HO와22 함께 6시간 배양하면 5-mCpG 부위의 상당한 탈메틸화가 발생하지만 HO22 배양 시간이 짧아지면 다른 후성유전학적 변화가 촉진되는 것으로 보입니다. 세포를22 HO로 30분 동안 처리하면 DNA 메틸트랜스퍼라제 1(DNMT1)을 어떤 종류의 산화적 DNA 손상 부위에 모집하기 위해 불일치 복구 단백질 이종이량체 MSH2-MSH6가 발생합니다.[137] 이는 이러한 위치에서 시토신의 메틸화(후성유전학적 변화)를 증가시킬 수 있습니다.
Jiang [138]등은 HEK 293 세포를 산화적 DNA 손상을 유발하는 약제(브롬산칼륨(KBrO3) 또는 크롬산칼륨(K2CrO4)로 치료했습니다. DNA 복구 효소 중합효소 베타가 산화된 구아닌에 국한되면서 산화 손상의 염기 절제 복구(BER)가 발생했습니다. 중합효소 베타는 산화 DNA 손상의 숏패치 BER에서 주요 인간 중합효소입니다. Jiang [138]등은 또한 중합효소 베타가 DNA 메틸트랜스퍼라제 단백질 DNMT3b를 BER 복구 부위로 모집한다는 것을 발견했습니다. 그 후 그들은 BRCA1 유전자의 프로모터 영역과 초기 전사 영역을 포함하는 DNA의 작은 영역에서 단일 뉴클레오티드 수준에서 메틸화 패턴을 평가했습니다. 브롬산염의 산화적 DNA 손상은 연구된 DNA 영역 내의 CpG 부위에서 DNA 메틸화 패턴(후성유전학적 변화 유발)을 조절했습니다. 처리되지 않은 세포에서 BRCA1 유전자의 -189, -134, -29, -19, +16 및 +19에 위치한 CpG는 메틸화된 시토신(여기서 번호는 전령 RNA 전사 시작 부위의 번호이고 음수는 상류 촉진체 영역의 뉴클레오티드를 나타냅니다)을 가지고 있습니다. 브롬산염 처리 유도 산화는 -189,[citation needed] -134, +16 및 +19에서 시토신 메틸화의 손실을 초래하는 동시에 DNA 복구가 허용된 후 -80, -55, -21 및 +8에 위치한 CpG에서 새로운 메틸화를 형성했습니다.
상동 재조합 복구는 후성유전학적 마커를 변경합니다.
DNA 이중 가닥 절단 부위에 DNA 메틸트랜스퍼라제 1(DNMT1)의 모집을 보고하는 기사는 적어도 4개입니다.[139][140][98][141] 이중 가닥 파손의 상동 재조합 복구(HR) 동안, DNMT1의 관여는 복구된 두 가닥의 DNA가 메틸화된 시토신의 다른 수준을 갖도록 합니다. 하나의 가닥은 복구된 이중 가닥 파손의 하류에 있는 약 21개의 CpG 부위에서 자주 메틸화됩니다. 다른 DNA 가닥은 이중 가닥 파손의 하류에서 이전에 메틸화된 약 6개의 CpG 부위에서 메틸화를 상실하고, 이중 가닥 파손의 상류에서 이전에 메틸화된 약 5개의 CpG 부위에서 메틸화를 상실합니다. 염색체가 복제되면 이전 절단 부위의 하류에서 심하게 메틸화되는 하나의 딸 염색체와 이전 절단 부위의 상류 및 하류 영역에서 메틸화되지 않는 하나의 딸 염색체가 생성됩니다. 이중 가닥이 끊어져서 깨진 유전자에 대해서는 자손 세포의 절반은 높은 수준으로 발현하고 나머지 절반은 해당 유전자의 발현이 억제됩니다. 이 세포들의 클론이 3년 동안 유지되었을 때, 그 기간 동안 새로운 메틸화 패턴이 유지되었습니다.[142]
유전체에 CRISPR 매개 상동성 유도 재조합 삽입이 있는 마우스에서 이중 가닥 파손 관련 삽입 내에서 CpG 부위의 증가된 메틸화가 많이 있었습니다.[143]
상동이 아닌 말단 결합은 후성유전학적 표지자의 변화를 일으킬 수 있습니다.
이중 가닥 파손의 비동형 말단 접합(NHEJ) 복구는 복구된 이중 가닥 파손의 하류에 있는 소수의 기존 사이토신 DNA 메틸화의 탈메틸화를 유발할 수 있습니다.[140] Allen et al.[144] 의 추가 연구는 세포에서 DNA 이중 가닥 분열의 NHEJ가 초기 이중 가닥 분열을 포함하는 유전자의 발현이 억제된 일부 자손 세포를 생성할 수 있음을 보여주었고 NHEJ 복구와 관련된 후생유전적 변화로 인해 해당 유전자의 발현이 높은 일부 자손 세포를 생성할 수 있음을 보여주었습니다. NHEJ 복구 후 유전자의 DNA 이중 가닥 파손을 억제하는 후생유전학적 변화의 빈도는 약 0.9%[98]일 수 있습니다.
진화
DNA 복구의 기본적인 과정은 원핵생물과 진핵생물, 심지어 박테리오파지(박테리아를 감염시키는 바이러스) 사이에서도 고도로 보존되어 있습니다. 그러나 더 복잡한 유전체를 가진 더 복잡한 유기체들은 그에 상응하여 더 복잡한 복구 메커니즘을 가지고 있습니다.[145] 관련 화학 반응을 촉매하는 많은 수의 단백질 구조 모티프의 능력은 진화 중 복구 메커니즘의 정교화에 중요한 역할을 했습니다. DNA 복구의 진화와 관련된 가설에 대한 매우 상세한 검토는 다음을 참조하십시오.[146]
화석 기록은 단세포 생물이 선캄브리아기의 어느 시점에 지구상에서 증식하기 시작했음을 나타내지만, 정확히 언제 인지할 수 있는 현대 생명체가 출현했는지는 불분명합니다. 핵산은 유전 정보를 암호화하는 유일하고 보편적인 수단이 되었으며, DNA 복구 메커니즘을 필요로 하며, 기본적인 형태는 공통 조상으로부터 현존하는 모든 생명체에 의해 유전됩니다. 광합성 생물로 인해 산소가 풍부한 지구 대기의 출현("산소 대재앙"으로 알려져 있음)과 산화적 인산화로 인해 세포 내에 잠재적으로 손상을 줄 수 있는 활성산소의 존재, 산화적 스트레스에 의해 유발되는 손상의 유형에 대항하기 위해 특별히 작용하는 DNA 복구 메커니즘의 진화가 필요했습니다. 그러나 이것이 발생한 메커니즘은 불분명합니다.[citation needed]
진화변화율
DNA 손상이 복구되지 않거나 오류가 발생하기 쉬운 메커니즘에 의해 복구되는 경우가 있으며 이로 인해 원래 서열이 변경됩니다. 이것이 발생하면 돌연변이가 세포 자손의 유전체로 전파될 수 있습니다. 그러한 사건이 생식 세포에서 발생하여 결국 배우자를 생성한다면, 돌연변이는 유기체의 자손에게 전달될 가능성이 있습니다. 특정 종(또는 특정 유전자)의 진화 속도는 돌연변이 속도의 함수입니다. 결과적으로 DNA 복구 메커니즘의 속도와 정확성은 진화적 변화 과정에 영향을 미칩니다.[147] DNA 손상 보호 및 복구는 유전자 조절 및 대립유전자의 재조합 및 선택에 의한 적응 속도에 영향을 미치지 않습니다. 반면, DNA 손상 복구 및 보호는 회복 불가능하고 유리하며, 코드 확장, 유전 가능한 돌연변이의 축적 속도에 영향을 미치며, 새로운 기능을 가진 유기체의 유전체 확장을 위한 진화 메커니즘을 늦춥니다. 진화 가능성과 돌연변이 복구 및 보호 사이의 긴장 관계는 추가 조사가 필요합니다.[citation needed]
기술
클러스터된 규칙적으로 간격을 둔 짧은 회문 반복(CRISPR-Cas9로 단축)이라는 이름의 기술이 2012년에 발견되었습니다. 이 새로운 기술은 분자생물학 훈련을 받은 사람이라면 누구나 특정 지점에서 DNA 손상을 유도한 다음 DNA 복구 메커니즘을 변경하여 새로운 유전자를 삽입함으로써 정확하게 모든 종의 유전자를 변경할 수 있습니다.[148] 다른 기술보다 저렴하고 효율적이며 정밀합니다. 크리스퍼-Cas9의 도움으로, DNA 서열의 일부를 제거, 추가, 또는 변경함으로써 과학자들에 의해 게놈의 일부를 편집할 수 있습니다.[citation needed]
참고 항목
- 가속노화병
- 노화 DNA
- 세포주기
- DNA 손상(자연발생)
- 노화에 대한 DNA 손상 이론
- DNA 복제
- 직접적인 DNA 손상
- 오류감지 및 수정
- 유전자치료
- 인간 미토콘드리아 유전학
- 간접 DNA 손상
- 수명연장
- 프로게리아
- 수리용 타이어
- 노쇠함
- SiDNA
- 돌연변이 연구를 통한 과학 저널 DNA 복구
참고문헌
- ^ "Nature Reviews Series: DNA damage". Nature Reviews Molecular Cell Biology. 5 July 2017. Retrieved 7 November 2018.
- ^ a b Lodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipursky SL, Darnell J (2004). Molecular Biology of the Cell (5th ed.). New York: WH Freeman. p. 963.
- ^ Browner WS, Kahn AJ, Ziv E, Reiner AP, Oshima J, Cawthon RM, et al. (December 2004). "The genetics of human longevity". The American Journal of Medicine. 117 (11): 851–60. CiteSeerX 10.1.1.556.6874. doi:10.1016/j.amjmed.2004.06.033. PMID 15589490.
- ^ Broad WJ (7 October 2015). "Nobel Prize in Chemistry Awarded to Tomas Lindahl, Paul Modrich and Aziz Sancar for DNA Studies". The New York Times. Retrieved 7 October 2015.
- ^ Staff (7 October 2015). "The Nobel Prize in Chemistry 2015 – DNA repair – providing chemical stability for life" (PDF). Nobel Prize. Retrieved 7 October 2015.
- ^ Roulston A, Marcellus RC, Branton PE (1999). "Viruses and apoptosis". Annual Review of Microbiology. 53: 577–628. doi:10.1146/annurev.micro.53.1.577. PMID 10547702.
- ^ Madigan MT, Martino JM (2006). Brock Biology of Microorganisms (11th ed.). Pearson. p. 136. ISBN 978-0-13-196893-6.
- ^ Ohta T, Tokishita SI, Mochizuki K, Kawase J, Sakahira M, Yamagata H (2006). "UV Sensitivity and Mutagenesis of the Extremely Thermophilic Eubacterium Thermus thermophilus HB27". Genes and Environment. 28 (2): 56–61. doi:10.3123/jemsge.28.56.
- ^ Tanaka T, Halicka HD, Huang X, Traganos F, Darzynkiewicz Z (September 2006). "Constitutive histone H2AX phosphorylation and ATM activation, the reporters of DNA damage by endogenous oxidants". Cell Cycle. 5 (17): 1940–45. doi:10.4161/cc.5.17.3191. PMC 3488278. PMID 16940754.
- ^ Braig M, Schmitt CA (March 2006). "Oncogene-induced senescence: putting the brakes on tumor development". Cancer Research. 66 (6): 2881–84. doi:10.1158/0008-5472.CAN-05-4006. PMID 16540631.
- ^ Lynch MD (February 2006). "How does cellular senescence prevent cancer?". DNA and Cell Biology. 25 (2): 69–78. doi:10.1089/dna.2006.25.69. PMID 16460230.
- ^ Campisi J, d'Adda di Fagagna F (September 2007). "Cellular senescence: when bad things happen to good cells". Nature Reviews. Molecular Cell Biology. 8 (9): 729–40. doi:10.1038/nrm2233. PMID 17667954. S2CID 15664931.
- ^ a b c Best BP (June 2009). "Nuclear DNA damage as a direct cause of aging" (PDF). Rejuvenation Research. 12 (3): 199–208. CiteSeerX 10.1.1.318.738. doi:10.1089/rej.2009.0847. PMID 19594328. Archived from the original (PDF) on 15 November 2017. Retrieved 29 September 2009.
- ^ Sancar A (June 2003). "Structure and function of DNA photolyase and cryptochrome blue-light photoreceptors". Chemical Reviews. 103 (6): 2203–37. doi:10.1021/cr0204348. PMID 12797829.
- ^ Lucas-Lledó JI, Lynch M (May 2009). "Evolution of mutation rates: phylogenomic analysis of the photolyase/cryptochrome family". Molecular Biology and Evolution. 26 (5): 1143–53. doi:10.1093/molbev/msp029. PMC 2668831. PMID 19228922.
- ^ a b c Watson JD, Baker TA, Bell SP, Gann A, Levine M, Losick R (2004). Molecular Biology of the Gene (5th ed.). Pearson Benjamin Cummings; CSHL Press. Ch. 9, 10. OCLC 936762772.
- ^ Volkert MR (1988). "Adaptive response of Escherichia coli to alkylation damage". Environmental and Molecular Mutagenesis. 11 (2): 241–55. doi:10.1002/em.2850110210. PMID 3278898. S2CID 24722637.
- ^ a b c Willey J, Sherwood L, Woolverton C (2014). Prescott's Microbiology. New York: McGraw Hill. p. 381. ISBN 978-0-07-3402-40-6.
- ^ Russell P (2018). i Genetics. Chennai: Pearson. p. 186. ISBN 978-93-325-7162-4.
- ^ a b c d Reardon JT, Sancar A (2006). "Purification and characterization of Escherichia coli and human nucleotide excision repair enzyme systems". DNA Repair, Part A. Methods in Enzymology. Vol. 408. pp. 189–213. doi:10.1016/S0076-6879(06)08012-8. ISBN 9780121828134. PMID 16793370.
- ^ Berg M, Tymoczko J, Stryer L (2012). Biochemistry 7th edition. New York: W.H. Freeman and Company. p. 840. ISBN 9781429229364.
- ^ Acharya PV (1971). "The isolation and partial characterization of age-correlated oligo-deoxyribo-ribonucleotides with covalently linked aspartyl-glutamyl polypeptides". Johns Hopkins Medical Journal. Supplement (1): 254–60. PMID 5055816.
- ^ Bjorksten J, Acharya PV, Ashman S, Wetlaufer DB (July 1971). "Gerogenic fractions in the tritiated rat". Journal of the American Geriatrics Society. 19 (7): 561–74. doi:10.1111/j.1532-5415.1971.tb02577.x. PMID 5106728. S2CID 33154242.
- ^ a b Liang L, Deng L, Chen Y, Li GC, Shao C, Tischfield JA (September 2005). "Modulation of DNA end joining by nuclear proteins". The Journal of Biological Chemistry. 280 (36): 31442–49. doi:10.1074/jbc.M503776200. PMID 16012167.
- ^ Wilson TE, Grawunder U, Lieber MR (July 1997). "Yeast DNA ligase IV mediates non-homologous DNA end joining". Nature. 388 (6641): 495–98. Bibcode:1997Natur.388..495W. doi:10.1038/41365. PMID 9242411. S2CID 4422938.
- ^ Moore JK, Haber JE (May 1996). "Cell cycle and genetic requirements of two pathways of nonhomologous end-joining repair of double-strand breaks in Saccharomyces cerevisiae". Molecular and Cellular Biology. 16 (5): 2164–73. doi:10.1128/mcb.16.5.2164. PMC 231204. PMID 8628283.
- ^ Boulton SJ, Jackson SP (September 1996). "Saccharomyces cerevisiae Ku70 potentiates illegitimate DNA double-strand break repair and serves as a barrier to error-prone DNA repair pathways". The EMBO Journal. 15 (18): 5093–103. doi:10.1002/j.1460-2075.1996.tb00890.x. PMC 452249. PMID 8890183.
- ^ Wilson TE, Lieber MR (August 1999). "Efficient processing of DNA ends during yeast nonhomologous end joining. Evidence for a DNA polymerase beta (Pol4)-dependent pathway". The Journal of Biological Chemistry. 274 (33): 23599–609. doi:10.1074/jbc.274.33.23599. PMID 10438542.
- ^ Budman J, Chu G (February 2005). "Processing of DNA for nonhomologous end-joining by cell-free extract". The EMBO Journal. 24 (4): 849–60. doi:10.1038/sj.emboj.7600563. PMC 549622. PMID 15692565.
- ^ Wang H, Perrault AR, Takeda Y, Qin W, Wang H, Iliakis G (September 2003). "Biochemical evidence for Ku-independent backup pathways of NHEJ". Nucleic Acids Research. 31 (18): 5377–88. doi:10.1093/nar/gkg728. PMC 203313. PMID 12954774.
- ^ Jung D, Alt FW (January 2004). "Unraveling V(D)J recombination; insights into gene regulation". Cell. 116 (2): 299–311. doi:10.1016/S0092-8674(04)00039-X. PMID 14744439. S2CID 16890458.
- ^ a b Truong LN, Li Y, Shi LZ, Hwang PY, He J, Wang H, et al. (May 2013). "Microhomology-mediated End Joining and Homologous Recombination share the initial end resection step to repair DNA double-strand breaks in mammalian cells". Proceedings of the National Academy of Sciences of the United States of America. 110 (19): 7720–25. Bibcode:2013PNAS..110.7720T. doi:10.1073/pnas.1213431110. PMC 3651503. PMID 23610439.
- ^ Sharma S, Javadekar SM, Pandey M, Srivastava M, Kumari R, Raghavan SC (March 2015). "Homology and enzymatic requirements of microhomology-dependent alternative end joining". Cell Death & Disease. 6 (3): e1697. doi:10.1038/cddis.2015.58. PMC 4385936. PMID 25789972.
- ^ Decottignies A (2013). "Alternative end-joining mechanisms: a historical perspective". Frontiers in Genetics. 4: 48. doi:10.3389/fgene.2013.00048. PMC 3613618. PMID 23565119.
- ^ Zahradka K, Slade D, Bailone A, Sommer S, Averbeck D, Petranovic M, et al. (October 2006). "Reassembly of shattered chromosomes in Deinococcus radiodurans". Nature. 443 (7111): 569–73. Bibcode:2006Natur.443..569Z. doi:10.1038/nature05160. PMID 17006450. S2CID 4412830.
- ^ Stenerlöw B, Karlson KH, Cooper B, Rydberg B. "열에 불안정한 부위를 포함하지 않고 포유동물 세포에서 신속한 DNA 이중 가닥 절단 측정: 비동종 말단 접합이 결핍된 세포에 대한 결과" Radiat Res. 2003년 4월 159(4):502–10. doi:10.1667/0033-7587(2003) 159[0502:mopdds2.0.co ;2] PMID 12643795
- ^ Waters LS, Minesinger BK, Wiltrout ME, D'Souza S, Woodruff RV, Walker GC (March 2009). "Eukaryotic translesion polymerases and their roles and regulation in DNA damage tolerance". Microbiology and Molecular Biology Reviews. 73 (1): 134–54. doi:10.1128/MMBR.00034-08. PMC 2650891. PMID 19258535.
- ^ Colis LC, Raychaudhury P, Basu AK (August 2008). "Mutational specificity of gamma-radiation-induced guanine-thymine and thymine-guanine intrastrand cross-links in mammalian cells and translesion synthesis past the guanine-thymine lesion by human DNA polymerase eta". Biochemistry. 47 (31): 8070–79. doi:10.1021/bi800529f. PMC 2646719. PMID 18616294.
- ^ Raychaudhury P, Basu AK (March 2011). "Genetic requirement for mutagenesis of the G[8,5-Me]T cross-link in Escherichia coli: DNA polymerases IV and V compete for error-prone bypass". Biochemistry. 50 (12): 2330–38. doi:10.1021/bi102064z. PMC 3062377. PMID 21302943.
- ^ "Translesion Synthesis". Research.chem.psu.edu. Archived from the original on 10 March 2012. Retrieved 14 August 2012.
- ^ Wang Z (July 2001). "Translesion synthesis by the UmuC family of DNA polymerases". Mutation Research. 486 (2): 59–70. doi:10.1016/S0921-8777(01)00089-1. PMID 11425512.
- ^ a b c Friedberg EC, Walker GC, Sied W, Wood RD, Schultz RA, Ellenberger T. (2006) DNA 복구 및 돌연변이 유발, 3부. ASM 프레스 2번째.
- ^ Liu B, Yip RK, Zhou Z (November 2012). "Chromatin remodeling, DNA damage repair and aging". Current Genomics. 13 (7): 533–47. doi:10.2174/138920212803251373. PMC 3468886. PMID 23633913.
- ^ Halicka HD, Zhao H, Podhorecka M, Traganos F, Darzynkiewicz Z (July 2009). "Cytometric detection of chromatin relaxation, an early reporter of DNA damage response". Cell Cycle. 8 (14): 2233–37. doi:10.4161/cc.8.14.8984. PMC 3856216. PMID 19502789.
- ^ a b c Sellou H, Lebeaupin T, Chapuis C, Smith R, Hegele A, Singh HR, et al. (December 2016). "The poly(ADP-ribose)-dependent chromatin remodeler Alc1 induces local chromatin relaxation upon DNA damage". Molecular Biology of the Cell. 27 (24): 3791–99. doi:10.1091/mbc.E16-05-0269. PMC 5170603. PMID 27733626.
- ^ a b Van Meter M, Simon M, Tombline G, May A, Morello TD, Hubbard BP, et al. (September 2016). "JNK Phosphorylates SIRT6 to Stimulate DNA Double-Strand Break Repair in Response to Oxidative Stress by Recruiting PARP1 to DNA Breaks". Cell Reports. 16 (10): 2641–50. doi:10.1016/j.celrep.2016.08.006. PMC 5089070. PMID 27568560.
- ^ a b Haince JF, McDonald D, Rodrigue A, Déry U, Masson JY, Hendzel MJ, Poirier GG (January 2008). "PARP1-dependent kinetics of recruitment of MRE11 and NBS1 proteins to multiple DNA damage sites". The Journal of Biological Chemistry. 283 (2): 1197–208. doi:10.1074/jbc.M706734200. PMID 18025084.
- ^ a b c Rogakou EP, Pilch DR, Orr AH, Ivanova VS, Bonner WM (March 1998). "DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139". The Journal of Biological Chemistry. 273 (10): 5858–68. doi:10.1074/jbc.273.10.5858. PMID 9488723.
- ^ Mailand N, Bekker-Jensen S, Faustrup H, Melander F, Bartek J, Lukas C, Lukas J (November 2007). "RNF8 ubiquitylates histones at DNA double-strand breaks and promotes assembly of repair proteins". Cell. 131 (5): 887–900. doi:10.1016/j.cell.2007.09.040. PMID 18001824. S2CID 14232192.
- ^ Luijsterburg MS, Acs K, Ackermann L, Wiegant WW, Bekker-Jensen S, Larsen DH, et al. (May 2012). "A new non-catalytic role for ubiquitin ligase RNF8 in unfolding higher-order chromatin structure". The EMBO Journal. 31 (11): 2511–27. doi:10.1038/emboj.2012.104. PMC 3365417. PMID 22531782.
- ^ a b Luijsterburg MS, Goedhart J, Moser J, Kool H, Geverts B, Houtsmuller AB, et al. (August 2007). "Dynamic in vivo interaction of DDB2 E3 ubiquitin ligase with UV-damaged DNA is independent of damage-recognition protein XPC". Journal of Cell Science. 120 (Pt 15): 2706–16. doi:10.1242/jcs.008367. PMID 17635991.
- ^ a b Pines A, Vrouwe MG, Marteijn JA, Typas D, Luijsterburg MS, Cansoy M, et al. (October 2012). "PARP1 promotes nucleotide excision repair through DDB2 stabilization and recruitment of ALC1". The Journal of Cell Biology. 199 (2): 235–49. doi:10.1083/jcb.201112132. PMC 3471223. PMID 23045548.
- ^ Jazayeri A, Falck J, Lukas C, Bartek J, Smith GC, Lukas J, Jackson SP (January 2006). "ATM- and cell cycle-dependent regulation of ATR in response to DNA double-strand breaks". Nature Cell Biology. 8 (1): 37–45. doi:10.1038/ncb1337. PMID 16327781. S2CID 9797133.
- ^ Bakkenist CJ, Kastan MB (January 2003). "DNA damage activates ATM through intermolecular autophosphorylation and dimer dissociation". Nature. 421 (6922): 499–506. Bibcode:2003Natur.421..499B. doi:10.1038/nature01368. PMID 12556884. S2CID 4403303.
- ^ Wei Q, Li L, Chen D (2007). DNA Repair, Genetic Instability, and Cancer. World Scientific. ISBN 978-981-270-014-8.[페이지 필요]
- ^ Schonthal AH (2004). Checkpoint Controls and Cancer. Humana Press. ISBN 978-1-58829-500-2.[페이지 필요]
- ^ Gartel AL, Tyner AL (June 2002). "The role of the cyclin-dependent kinase inhibitor p21 in apoptosis". Molecular Cancer Therapeutics. 1 (8): 639–49. PMID 12479224.
- ^ Janion C (2001). "Some aspects of the SOS response system--a critical survey". Acta Biochimica Polonica. 48 (3): 599–610. doi:10.18388/abp.2001_3894. PMID 11833768.
- ^ a b Erill I, Campoy S, Barbé J (November 2007). "Aeons of distress: an evolutionary perspective on the bacterial SOS response". FEMS Microbiology Reviews. 31 (6): 637–56. doi:10.1111/j.1574-6976.2007.00082.x. PMID 17883408.
- ^ Schlacher K, Pham P, Cox MM, Goodman MF (February 2006). "Roles of DNA polymerase V and RecA protein in SOS damage-induced mutation". Chemical Reviews. 106 (2): 406–19. doi:10.1021/cr0404951. PMID 16464012.
- ^ Fry RC, Begley TJ, Samson LD (2004). "Genome-wide responses to DNA-damaging agents". Annual Review of Microbiology. 59: 357–77. doi:10.1146/annurev.micro.59.031805.133658. PMID 16153173.
- ^ Espejel S, Martín M, Klatt P, Martín-Caballero J, Flores JM, Blasco MA (May 2004). "Shorter telomeres, accelerated ageing and increased lymphoma in DNA-PKcs-deficient mice". EMBO Reports. 5 (5): 503–09. doi:10.1038/sj.embor.7400127. PMC 1299048. PMID 15105825.
- ^ de Boer J, Andressoo JO, de Wit J, Huijmans J, Beems RB, van Steeg H, et al. (May 2002). "Premature aging in mice deficient in DNA repair and transcription". Science. 296 (5571): 1276–79. Bibcode:2002Sci...296.1276D. doi:10.1126/science.1070174. PMID 11950998. S2CID 41930529.
- ^ Dollé ME, Busuttil RA, Garcia AM, Wijnhoven S, van Drunen E, Niedernhofer LJ, et al. (April 2006). "Increased genomic instability is not a prerequisite for shortened lifespan in DNA repair deficient mice". Mutation Research. 596 (1–2): 22–35. doi:10.1016/j.mrfmmm.2005.11.008. PMID 16472827.
- ^ a b c MacRae SL, Croken MM, Calder RB, Aliper A, Milholand B, White RR, Zavoronkov A, Gladyshev VN, Seluanov A, Gorbunova V, Zhang ZD, Vijg J (2015). "수명 차이가 극심한 종의 DNA 복구" Aging. 7 (12): 1171–84. doi:10.18632/aging.100866. PMC 4712340. PMID 26729707
- ^ Kobayashi Y, Narumi I, Satoh K, Funayama T, Kikuchi M, Kitayama S, Watanabe H (November 2004). "Radiation response mechanisms of the extremely radioresistant bacterium Deinococcus radiodurans". Uchu Seibutsu Kagaku. 18 (3): 134–35. PMID 15858357.
- ^ Spindler SR (September 2005). "Rapid and reversible induction of the longevity, anticancer and genomic effects of caloric restriction". Mechanisms of Ageing and Development. 126 (9): 960–66. doi:10.1016/j.mad.2005.03.016. PMID 15927235. S2CID 7067036.
- ^ Halicka HD, Zhao H, Li J, Lee YS, Hsieh TC, Wu JM, Darzynkiewicz Z (December 2012). "Potential anti-aging agents suppress the level of constitutive mTOR- and DNA damage- signaling". Aging. 4 (12): 952–65. doi:10.18632/aging.100521. PMC 3615161. PMID 23363784.
- ^ Tissenbaum HA, Guarente L (March 2001). "Increased dosage of a sir-2 gene extends lifespan in Caenorhabditis elegans". Nature. 410 (6825): 227–30. Bibcode:2001Natur.410..227T. doi:10.1038/35065638. PMID 11242085. S2CID 4356885.
- ^ Cohen HY, Miller C, Bitterman KJ, Wall NR, Hekking B, Kessler B, et al. (July 2004). "Calorie restriction promotes mammalian cell survival by inducing the SIRT1 deacetylase". Science. 305 (5682): 390–92. Bibcode:2004Sci...305..390C. doi:10.1126/science.1099196. PMID 15205477. S2CID 33503081.
- ^ Cabelof DC, Yanamadala S, Raffoul JJ, Guo Z, Soofi A, Heydari AR (March 2003). "Caloric restriction promotes genomic stability by induction of base excision repair and reversal of its age-related decline". DNA Repair. 2 (3): 295–307. doi:10.1016/S1568-7864(02)00219-7. PMID 12547392.
- ^ Stuart JA, Karahalil B, Hogue BA, Souza-Pinto NC, Bohr VA (March 2004). "Mitochondrial and nuclear DNA base excision repair are affected differently by caloric restriction". FASEB Journal. 18 (3): 595–97. doi:10.1096/fj.03-0890fje. PMID 14734635. S2CID 43118901.
- ^ Walker DW, McColl G, Jenkins NL, Harris J, Lithgow GJ (May 2000). "Evolution of lifespan in C. elegans". Nature. 405 (6784): 296–97. doi:10.1038/35012693. PMID 10830948. S2CID 4402039.
- ^ Johnson G (28 December 2010). "Unearthing Prehistoric Tumors, and Debate". The New York Times.
If we lived long enough, sooner or later we all would get cancer.
- ^ Alberts B, Johnson A, Lewis J, et al. (2002). "The Preventable Causes of Cancer". Molecular biology of the cell (4th ed.). New York: Garland Science. ISBN 978-0-8153-4072-0.
A certain irreducible background incidence of cancer is to be expected regardless of circumstances: mutations can never be absolutely avoided, because they are an inescapable consequence of fundamental limitations on the accuracy of DNA replication, as discussed in Chapter 5. If a human could live long enough, it is inevitable that at least one of his or her cells would eventually accumulate a set of mutations sufficient for cancer to develop.
- ^ Friedenson B (August 2007). "The BRCA1/2 pathway prevents hematologic cancers in addition to breast and ovarian cancers". BMC Cancer. 7: 152. doi:10.1186/1471-2407-7-152. PMC 1959234. PMID 17683622.
- ^ Gavande NS, VanderVere-Carozza PS, Hinshaw HD, Jalal SI, Sears CR, Pawelczak KS, Turchi JJ (April 2016). "DNA repair targeted therapy: The past or future of cancer treatment?". Pharmacology & Therapeutics. 160: 65–83. doi:10.1016/j.pharmthera.2016.02.003. PMC 4811676. PMID 26896565.
- ^ Bryant HE, Schultz N, Thomas HD, Parker KM, Flower D, Lopez E, et al. (April 2005). "Specific killing of BRCA2-deficient tumours with inhibitors of poly(ADP-ribose) polymerase". Nature. 434 (7035): 913–17. Bibcode:2005Natur.434..913B. doi:10.1038/nature03443. PMID 15829966. S2CID 4391043.
- ^ Goldstein M, Kastan MB (2015). "The DNA damage response: implications for tumor responses to radiation and chemotherapy". Annual Review of Medicine. 66: 129–43. doi:10.1146/annurev-med-081313-121208. PMID 25423595.
- ^ a b Jeggo PA, Pearl LH, Carr AM (January 2016). "DNA repair, genome stability and cancer: a historical perspective" (PDF). Nature Reviews. Cancer. 16 (1): 35–42. doi:10.1038/nrc.2015.4. PMID 26667849. S2CID 14941857.
- ^ Bartkova J, Horejsí Z, Koed K, Krämer A, Tort F, Zieger K, et al. (April 2005). "DNA damage response as a candidate anti-cancer barrier in early human tumorigenesis". Nature. 434 (7035): 864–70. Bibcode:2005Natur.434..864B. doi:10.1038/nature03482. PMID 15829956. S2CID 4398393.
- ^ a b Bartkova J, Rezaei N, Liontos M, Karakaidos P, Kletsas D, Issaeva N, et al. (November 2006). "Oncogene-induced senescence is part of the tumorigenesis barrier imposed by DNA damage checkpoints". Nature. 444 (7119): 633–37. Bibcode:2006Natur.444..633B. doi:10.1038/nature05268. PMID 17136093. S2CID 4406956.
- ^ Gaillard H, García-Muse T, Aguilera A (May 2015). "Replication stress and cancer". Nature Reviews. Cancer. 15 (5): 276–89. doi:10.1038/nrc3916. hdl:10261/123721. PMID 25907220. S2CID 11342123.
- ^ Halazonetis TD, Gorgoulis VG, Bartek J (March 2008). "An oncogene-induced DNA damage model for cancer development". Science. 319 (5868): 1352–55. Bibcode:2008Sci...319.1352H. doi:10.1126/science.1140735. PMID 18323444. S2CID 16426080.
- ^ de Boer J, Hoeijmakers JH (March 2000). "Nucleotide excision repair and human syndromes" (PDF). Carcinogenesis. 21 (3): 453–60. doi:10.1093/carcin/21.3.453. PMID 10688865.
- ^ Broustas CG, Lieberman HB (February 2014). "DNA damage response genes and the development of cancer metastasis". Radiation Research. 181 (2): 111–30. Bibcode:2014RadR..181..111B. doi:10.1667/RR13515.1. PMC 4064942. PMID 24397478.
- ^ Zhang P, Wang J, Gao W, Yuan BZ, Rogers J, Reed E (May 2004). "CHK2 kinase expression is down-regulated due to promoter methylation in non-small cell lung cancer". Molecular Cancer. 3 (4): 14. doi:10.1186/1476-4598-3-14. PMC 419366. PMID 15125777.
- ^ Baylin SB, Ohm JE (February 2006). "Epigenetic gene silencing in cancer – a mechanism for early oncogenic pathway addiction?". Nature Reviews. Cancer. 6 (2): 107–16. doi:10.1038/nrc1799. PMID 16491070. S2CID 2514545.
- ^ Kanwal R, Gupta S (April 2012). "Epigenetic modifications in cancer". Clinical Genetics. 81 (4): 303–11. doi:10.1111/j.1399-0004.2011.01809.x. PMC 3590802. PMID 22082348.
- ^ Baldassarre G, Battista S, Belletti B, Thakur S, Pentimalli F, Trapasso F, et al. (April 2003). "Negative regulation of BRCA1 gene expression by HMGA1 proteins accounts for the reduced BRCA1 protein levels in sporadic breast carcinoma". Molecular and Cellular Biology. 23 (7): 2225–38. doi:10.1128/MCB.23.7.2225-2238.2003. PMC 150734. PMID 12640109.
- ^ Jacinto FV, Esteller M (July 2007). "Mutator pathways unleashed by epigenetic silencing in human cancer". Mutagenesis. 22 (4): 247–53. doi:10.1093/mutage/gem009. PMID 17412712.
- ^ Lahtz C, Pfeifer GP (February 2011). "Epigenetic changes of DNA repair genes in cancer". Journal of Molecular Cell Biology. 3 (1): 51–58. doi:10.1093/jmcb/mjq053. PMC 3030973. PMID 21278452. 암에서 DNA 복구 유전자의 후성유전학적 변화
- ^ Bernstein C, Nfonsam V, Prasad AR, Bernstein H (March 2013). "Epigenetic field defects in progression to cancer". World Journal of Gastrointestinal Oncology. 5 (3): 43–49. doi:10.4251/wjgo.v5.i3.43. PMC 3648662. PMID 23671730.
- ^ Narayanan L, Fritzell JA, Baker SM, Liskay RM, Glazer PM (April 1997). "Elevated levels of mutation in multiple tissues of mice deficient in the DNA mismatch repair gene Pms2". Proceedings of the National Academy of Sciences of the United States of America. 94 (7): 3122–27. Bibcode:1997PNAS...94.3122N. doi:10.1073/pnas.94.7.3122. PMC 20332. PMID 9096356.
- ^ Hegan DC, Narayanan L, Jirik FR, Edelmann W, Liskay RM, Glazer PM (December 2006). "Differing patterns of genetic instability in mice deficient in the mismatch repair genes Pms2, Mlh1, Msh2, Msh3 and Msh6". Carcinogenesis. 27 (12): 2402–08. doi:10.1093/carcin/bgl079. PMC 2612936. PMID 16728433.
- ^ Tutt AN, van Oostrom CT, Ross GM, van Steeg H, Ashworth A (March 2002). "Disruption of Brca2 increases the spontaneous mutation rate in vivo: synergism with ionizing radiation". EMBO Reports. 3 (3): 255–60. doi:10.1093/embo-reports/kvf037. PMC 1084010. PMID 11850397.
- ^ German J (March 1969). "Bloom's syndrome. I. Genetical and clinical observations in the first twenty-seven patients". American Journal of Human Genetics. 21 (2): 196–227. PMC 1706430. PMID 5770175.
- ^ a b c O'Hagan HM, Mohammad HP, Baylin SB (August 2008). "Double strand breaks can initiate gene silencing and SIRT1-dependent onset of DNA methylation in an exogenous promoter CpG island". PLOS Genetics. 4 (8): e1000155. doi:10.1371/journal.pgen.1000155. PMC 2491723. PMID 18704159.
- ^ Cuozzo C, Porcellini A, Angrisano T, Morano A, Lee B, Di Pardo A, et al. (July 2007). "DNA damage, homology-directed repair, and DNA methylation". PLOS Genetics. 3 (7): e110. doi:10.1371/journal.pgen.0030110. PMC 1913100. PMID 17616978.
- ^ Malkin D (April 2011). "Li-fraumeni syndrome". Genes & Cancer. 2 (4): 475–84. doi:10.1177/1947601911413466. PMC 3135649. PMID 21779515.
- ^ Fearon ER (November 1997). "Human cancer syndromes: clues to the origin and nature of cancer". Science. 278 (5340): 1043–50. Bibcode:1997Sci...278.1043F. doi:10.1126/science.278.5340.1043. PMID 9353177.
- ^ Halford S, Rowan A, Sawyer E, Talbot I, Tomlinson I (June 2005). "O(6)-methylguanine methyltransferase in colorectal cancers: detection of mutations, loss of expression, and weak association with G:C>A:T transitions". Gut. 54 (6): 797–802. doi:10.1136/gut.2004.059535. PMC 1774551. PMID 15888787.
- ^ Shen L, Kondo Y, Rosner GL, Xiao L, Hernandez NS, Vilaythong J, et al. (September 2005). "MGMT promoter methylation and field defect in sporadic colorectal cancer". Journal of the National Cancer Institute. 97 (18): 1330–38. doi:10.1093/jnci/dji275. PMID 16174854.
- ^ Psofaki V, Kalogera C, Tzambouras N, Stephanou D, Tsianos E, Seferiadis K, Kolios G (July 2010). "Promoter methylation status of hMLH1, MGMT, and CDKN2A/p16 in colorectal adenomas". World Journal of Gastroenterology. 16 (28): 3553–60. doi:10.3748/wjg.v16.i28.3553. PMC 2909555. PMID 20653064.
- ^ Lee KH, Lee JS, Nam JH, Choi C, Lee MC, Park CS, et al. (October 2011). "Promoter methylation status of hMLH1, hMSH2, and MGMT genes in colorectal cancer associated with adenoma-carcinoma sequence". Langenbeck's Archives of Surgery. 396 (7): 1017–26. doi:10.1007/s00423-011-0812-9. PMID 21706233. S2CID 8069716.
- ^ Amatu A, Sartore-Bianchi A, Moutinho C, Belotti A, Bencardino K, Chirico G, et al. (April 2013). "Promoter CpG island hypermethylation of the DNA repair enzyme MGMT predicts clinical response to dacarbazine in a phase II study for metastatic colorectal cancer". Clinical Cancer Research. 19 (8): 2265–72. doi:10.1158/1078-0432.CCR-12-3518. PMID 23422094.
- ^ Mokarram P, Zamani M, Kavousipour S, Naghibalhossaini F, Irajie C, Moradi Sarabi M, Hosseini SV (May 2013). "Different patterns of DNA methylation of the two distinct O6-methylguanine-DNA methyltransferase (O6-MGMT) promoter regions in colorectal cancer". Molecular Biology Reports. 40 (5): 3851–57. doi:10.1007/s11033-012-2465-3. PMID 23271133. S2CID 18733871.
- ^ Truninger K, Menigatti M, Luz J, Russell A, Haider R, Gebbers JO, et al. (May 2005). "Immunohistochemical analysis reveals high frequency of PMS2 defects in colorectal cancer". Gastroenterology. 128 (5): 1160–71. doi:10.1053/j.gastro.2005.01.056. PMID 15887099.
- ^ Valeri N, Gasparini P, Fabbri M, Braconi C, Veronese A, Lovat F, et al. (April 2010). "Modulation of mismatch repair and genomic stability by miR-155". Proceedings of the National Academy of Sciences of the United States of America. 107 (15): 6982–87. Bibcode:2010PNAS..107.6982V. doi:10.1073/pnas.1002472107. PMC 2872463. PMID 20351277.
- ^ Facista A, Nguyen H, Lewis C, Prasad AR, Ramsey L, Zaitlin B, et al. (April 2012). "Deficient expression of DNA repair enzymes in early progression to sporadic colon cancer". Genome Integrity. 3 (1): 3. doi:10.1186/2041-9414-3-3. PMC 3351028. PMID 22494821.
- ^ 인간 DNA 복구 유전자, 2014년 4월 15일, 텍사스 대학교 MD Anderson Cancer Center
- ^ Jin B, Robertson KD (2013). "DNA Methyltransferases, DNA Damage Repair, and Cancer". Epigenetic Alterations in Oncogenesis. Advances in Experimental Medicine and Biology. Vol. 754. pp. 3–29. doi:10.1007/978-1-4419-9967-2_1. ISBN 978-1-4419-9966-5. PMC 3707278. PMID 22956494.
- ^ Krishnan K, Steptoe AL, Martin HC, Wani S, Nones K, Waddell N, et al. (February 2013). "MicroRNA-182-5p targets a network of genes involved in DNA repair". RNA. 19 (2): 230–42. doi:10.1261/rna.034926.112. PMC 3543090. PMID 23249749.
- ^ Chaisaingmongkol J, Popanda O, Warta R, Dyckhoff G, Herpel E, Geiselhart L, et al. (December 2012). "Epigenetic screen of human DNA repair genes identifies aberrant promoter methylation of NEIL1 in head and neck squamous cell carcinoma". Oncogene. 31 (49): 5108–16. doi:10.1038/onc.2011.660. PMID 22286769.
- ^ Singh P, Yang M, Dai H, Yu D, Huang Q, Tan W, et al. (November 2008). "Overexpression and hypomethylation of flap endonuclease 1 gene in breast and other cancers". Molecular Cancer Research. 6 (11): 1710–17. doi:10.1158/1541-7786.MCR-08-0269. PMC 2948671. PMID 19010819.
- ^ Lam JS, Seligson DB, Yu H, Li A, Eeva M, Pantuck AJ, et al. (August 2006). "Flap endonuclease 1 is overexpressed in prostate cancer and is associated with a high Gleason score". BJU International. 98 (2): 445–51. doi:10.1111/j.1464-410X.2006.06224.x. PMID 16879693. S2CID 22165252.
- ^ Kim JM, Sohn HY, Yoon SY, Oh JH, Yang JO, Kim JH, et al. (January 2005). "Identification of gastric cancer-related genes using a cDNA microarray containing novel expressed sequence tags expressed in gastric cancer cells". Clinical Cancer Research. 11 (2 Pt 1): 473–82. doi:10.1158/1078-0432.473.11.2. PMID 15701830.
- ^ Wang K, Xie C, Chen D (May 2014). "Flap endonuclease 1 is a promising candidate biomarker in gastric cancer and is involved in cell proliferation and apoptosis". International Journal of Molecular Medicine. 33 (5): 1268–74. doi:10.3892/ijmm.2014.1682. PMID 24590400.
- ^ Krause A, Combaret V, Iacono I, Lacroix B, Compagnon C, Bergeron C, et al. (July 2005). "Genome-wide analysis of gene expression in neuroblastomas detected by mass screening" (PDF). Cancer Letters. 225 (1): 111–20. doi:10.1016/j.canlet.2004.10.035. PMID 15922863. S2CID 44644467.
- ^ Iacobuzio-Donahue CA, Maitra A, Olsen M, Lowe AW, van Heek NT, Rosty C, et al. (April 2003). "Exploration of global gene expression patterns in pancreatic adenocarcinoma using cDNA microarrays". The American Journal of Pathology. 162 (4): 1151–62. doi:10.1016/S0002-9440(10)63911-9. PMC 1851213. PMID 12651607.
- ^ Sato M, Girard L, Sekine I, Sunaga N, Ramirez RD, Kamibayashi C, Minna JD (October 2003). "Increased expression and no mutation of the Flap endonuclease (FEN1) gene in human lung cancer". Oncogene. 22 (46): 7243–46. doi:10.1038/sj.onc.1206977. PMID 14562054.
- ^ Bi FF, Li D, Yang Q (2013). "Hypomethylation of ETS transcription factor binding sites and upregulation of PARP1 expression in endometrial cancer". BioMed Research International. 2013: 946268. doi:10.1155/2013/946268. PMC 3666359. PMID 23762867.
- ^ Bi FF, Li D, Yang Q (February 2013). "Promoter hypomethylation, especially around the E26 transformation-specific motif, and increased expression of poly (ADP-ribose) polymerase 1 in BRCA-mutated serous ovarian cancer". BMC Cancer. 13: 90. doi:10.1186/1471-2407-13-90. PMC 3599366. PMID 23442605.
- ^ Supek F, Lehner B (May 2015). "Differential DNA mismatch repair underlies mutation rate variation across the human genome". Nature. 521 (7550): 81–84. Bibcode:2015Natur.521...81S. doi:10.1038/nature14173. PMC 4425546. PMID 25707793.
- ^ a b Zheng CL, Wang NJ, Chung J, Moslehi H, Sanborn JZ, Hur JS, et al. (November 2014). "Transcription restores DNA repair to heterochromatin, determining regional mutation rates in cancer genomes". Cell Reports. 9 (4): 1228–34. doi:10.1016/j.celrep.2014.10.031. PMC 4254608. PMID 25456125.
- ^ Li F, Mao G, Tong D, Huang J, Gu L, Yang W, Li GM (April 2013). "The histone mark H3K36me3 regulates human DNA mismatch repair through its interaction with MutSα". Cell. 153 (3): 590–600. doi:10.1016/j.cell.2013.03.025. PMC 3641580. PMID 23622243.
- ^ Supek F, Lehner B (July 2017). "Clustered Mutation Signatures Reveal that Error-Prone DNA Repair Targets Mutations to Active Genes". Cell. 170 (3): 534–547.e23. doi:10.1016/j.cell.2017.07.003. hdl:10230/35343. PMID 28753428.
- ^ Polak P, Lawrence MS, Haugen E, Stoletzki N, Stojanov P, Thurman RE, et al. (January 2014). "Reduced local mutation density in regulatory DNA of cancer genomes is linked to DNA repair". Nature Biotechnology. 32 (1): 71–75. doi:10.1038/nbt.2778. PMC 4116484. PMID 24336318.
- ^ Swenberg JA, Lu K, Moeller BC, Gao L, Upton PB, Nakamura J, Starr TB (March 2011). "Endogenous versus exogenous DNA adducts: their role in carcinogenesis, epidemiology, and risk assessment". Toxicol Sci. 120 (Suppl 1): S130–45. doi:10.1093/toxsci/kfq371. PMC 3043087. PMID 21163908.
- ^ a b Hamilton ML, Guo Z, Fuller CD, Van Remmen H, Ward WF, Austad SN, Troyer DA, Thompson I, Richardson A (May 2001). "A reliable assessment of 8-oxo-2-deoxyguanosine levels in nuclear and mitochondrial DNA using the sodium iodide method to isolate DNA". Nucleic Acids Res. 29 (10): 2117–26. doi:10.1093/nar/29.10.2117. PMC 55450. PMID 11353081.
- ^ Ming X, Matter B, Song M, Veliath E, Shanley R, Jones R, Tretyakova N (March 2014). "Mapping structurally defined guanine oxidation products along DNA duplexes: influence of local sequence context and endogenous cytosine methylation". J Am Chem Soc. 136 (11): 4223–35. doi:10.1021/ja411636j. PMC 3985951. PMID 24571128.
- ^ a b c Zhou X, Zhuang Z, Wang W, He L, Wu H, Cao Y, Pan F, Zhao J, Hu Z, Sekhar C, Guo Z (September 2016). "OGG1 is essential in oxidative stress-induced DNA demethylation". Cell Signal. 28 (9): 1163–1171. doi:10.1016/j.cellsig.2016.05.021. PMID 27251462.
- ^ Poetsch AR (2020). "The genomics of oxidative DNA damage, repair, and resulting mutagenesis". Comput Struct Biotechnol J. 18: 207–219. doi:10.1016/j.csbj.2019.12.013. PMC 6974700. PMID 31993111.
- ^ D'Augustin O, Huet S, Campalans A, Radicella JP (November 2020). "Lost in the Crowd: How Does Human 8-Oxoguanine DNA Glycosylase 1 (OGG1) Find 8-Oxoguanine in the Genome?". Int J Mol Sci. 21 (21): 8360. doi:10.3390/ijms21218360. PMC 7664663. PMID 33171795.
- ^ Lan L, Nakajima S, Oohata Y, Takao M, Okano S, Masutani M, Wilson SH, Yasui A (September 2004). "In situ analysis of repair processes for oxidative DNA damage in mammalian cells". Proc Natl Acad Sci U S A. 101 (38): 13738–43. Bibcode:2004PNAS..10113738L. doi:10.1073/pnas.0406048101. PMC 518826. PMID 15365186.
- ^ Maeder ML, Angstman JF, Richardson ME, Linder SJ, Cascio VM, Tsai SQ, Ho QH, Sander JD, Reyon D, Bernstein BE, Costello JF, Wilkinson MF, Joung JK (December 2013). "Targeted DNA demethylation and activation of endogenous genes using programmable TALE-TET1 fusion proteins". Nat. Biotechnol. 31 (12): 1137–42. doi:10.1038/nbt.2726. PMC 3858462. PMID 24108092.
- ^ Ding N, Bonham EM, Hannon BE, Amick TR, Baylin SB, O'Hagan HM (June 2016). "Mismatch repair proteins recruit DNA methyltransferase 1 to sites of oxidative DNA damage". J Mol Cell Biol. 8 (3): 244–54. doi:10.1093/jmcb/mjv050. PMC 4937888. PMID 26186941.
- ^ a b Jiang Z, Lai Y, Beaver JM, Tsegay PS, Zhao ML, Horton JK, Zamora M, Rein HL, Miralles F, Shaver M, Hutcheson JD, Agoulnik I, Wilson SH, Liu Y (January 2020). "Oxidative DNA Damage Modulates DNA Methylation Pattern in Human Breast Cancer 1 (BRCA1) Gene via the Crosstalk between DNA Polymerase β and a de novo DNA Methyltransferase". Cells. 9 (1): 225. doi:10.3390/cells9010225. PMC 7016758. PMID 31963223.
- ^ Mortusewicz O, Schermelleh L, Walter J, Cardoso MC, Leonhardt H (June 2005). "Recruitment of DNA methyltransferase I to DNA repair sites". Proc Natl Acad Sci U S A. 102 (25): 8905–9. Bibcode:2005PNAS..102.8905M. doi:10.1073/pnas.0501034102. PMC 1157029. PMID 15956212.
- ^ a b Cuozzo C, Porcellini A, Angrisano T, Morano A, Lee B, Di Pardo A, Messina S, Iuliano R, Fusco A, Santillo MR, Muller MT, Chiariotti L, Gottesman ME, Avvedimento EV (July 2007). "DNA damage, homology-directed repair, and DNA methylation". PLOS Genet. 3 (7): e110. doi:10.1371/journal.pgen.0030110. PMC 1913100. PMID 17616978.
- ^ Ha K, Lee GE, Palii SS, Brown KD, Takeda Y, Liu K, Bhalla KN, Robertson KD (January 2011). "Rapid and transient recruitment of DNMT1 to DNA double-strand breaks is mediated by its interaction with multiple components of the DNA damage response machinery". Hum Mol Genet. 20 (1): 126–40. doi:10.1093/hmg/ddq451. PMC 3000680. PMID 20940144.
- ^ Russo G, Landi R, Pezone A, Morano A, Zuchegna C, Romano A, Muller MT, Gottesman ME, Porcellini A, Avvedimento EV (September 2016). "DNA damage and Repair Modify DNA methylation and Chromatin Domain of the Targeted Locus: Mechanism of allele methylation polymorphism". Sci Rep. 6: 33222. Bibcode:2016NatSR...633222R. doi:10.1038/srep33222. PMC 5024116. PMID 27629060.
- ^ Farris MH, Texter PA, Mora AA, Wiles MV, Mac Garrigle EF, Klaus SA, Rosfjord K (December 2020). "Detection of CRISPR-mediated genome modifications through altered methylation patterns of CpG islands". BMC Genomics. 21 (1): 856. doi:10.1186/s12864-020-07233-2. PMC 7709351. PMID 33267773.
- ^ Allen B, Pezone A, Porcellini A, Muller MT, Masternak MM (June 2017). "Non-homologous end joining induced alterations in DNA methylation: A source of permanent epigenetic change". Oncotarget. 8 (25): 40359–40372. doi:10.18632/oncotarget.16122. PMC 5522286. PMID 28423717.
- ^ Cromie GA, Connelly JC, Leach DR (December 2001). "Recombination at double-strand breaks and DNA ends: conserved mechanisms from phage to humans". Molecular Cell. 8 (6): 1163–74. doi:10.1016/S1097-2765(01)00419-1. PMID 11779493.
- ^ O'Brien PJ (February 2006). "Catalytic promiscuity and the divergent evolution of DNA repair enzymes". Chemical Reviews. 106 (2): 720–52. doi:10.1021/cr040481v. PMID 16464022.
- ^ Maresca B, Schwartz JH (January 2006). "Sudden origins: a general mechanism of evolution based on stress protein concentration and rapid environmental change". The Anatomical Record Part B: The New Anatomist. 289 (1): 38–46. doi:10.1002/ar.b.20089. PMID 16437551.
- ^ "CRISPR 유전자 편집 도구는 과학자들을 흥분시키기도 했지만 긴장시키기도 했습니다" CBC 뉴스 저자 켈리 크로우. 2015년 11월 30일.
외부 링크
| 라이브러리 리소스 정보 DNA 복구 |
 Wikimedia Commons의 DNA 복구 관련 매체
Wikimedia Commons의 DNA 복구 관련 매체- 로스웰 파크 암 연구소 DNA 복구 강의
- 인간 DNA 복구 유전자의 포괄적인 목록
- 일부 DNA 복구 효소의 3D 구조
- 인간의 DNA 복구 질환
- DNA 복구 특별 관심 그룹
- 2018년 2월 12일 웨이백 기계에서 DNA 수리 보관
- DNA 손상 및 DNA 복구
- 분절 프로게리아
- DNA 손상 복구; 좋은 것, 나쁜 것, 못생긴 것
- 중금속 노출이 이중 가닥 파손 DNA 복구 결과에 영향을 미칩니다.


