전압 의존 음이온 채널
Voltage-dependent anion channel| 진핵포린 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
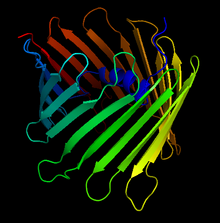 인간 전압 의존 음이온 채널의 결정구조.화살표는 특징적인 베타-바렐을 형성하는 대타렐 베타 시트를 나타낸다. | |||||||||
| 식별자 | |||||||||
| 기호 | 포린_3 | ||||||||
| Pfam | PF01459 | ||||||||
| 인터프로 | IPR001925 | ||||||||
| 프로사이트 | PDOC00483 | ||||||||
| TCDB | 1.B.8 | ||||||||
| OPM 슈퍼 패밀리 | 189 | ||||||||
| OPM단백질 | 3emn | ||||||||
| CDD | cd07306 | ||||||||
| |||||||||
전압 의존형 음이온 채널 또는 미토콘드리아 포린(mitochondrial porin)은 외부 미토콘드리아 막에 위치한 포린 이온 채널의 일종이다.[1][2]이 채널이 세포표면막에 표현되는지 아닌지에 대한 논쟁이 있다.[3][4][5]
진핵생물의 외측 미토콘드리아 막의 이 주요 단백질은 전압 의존형 음이온 선택적 채널(VDAC)을 형성하여 소수분자에 대한 일반 확산 공극으로 작용한다.[6][7][8][9]채널은 낮은 또는 0의 멤브레인 전위일 때 개방 정합성을 채택하고 30~40mV 이상의 전위일 때 폐쇄 정합성을 채택한다.VDAC는 미토콘드리아와 시토솔 사이의 이온과 분자의 교환을 촉진하고 다른 단백질과 작은 분자와의 상호작용에 의해 조절된다.[10]
구조
이 단백질은 약 280개의 아미노산을 함유하고 있으며 미토콘드리아 외막에 걸쳐 있는 베타 배럴을 형성하고 있다.[11][12]
1976년 발견 이후 VDAC 단백질의 광범위한 기능과 구조 분석이 수행되었다.모공에서 두드러진 특징이 나타났다. 평면 지질 빌레이어로 재구성할 때 대사물 플럭스가 높은 음이온 선택 고전도 상태와 대사물 통로가 제한된 양이온 선택 저전도 상태 사이에 전압 의존적 스위치가 있다.
최초 발견 후 30여 년이 지난 2008년 VDAC-1의 3개 독립 구조 프로젝트가 완료됐다.첫 번째는 다차원 NMR 분광법에 의해 해결되었다.두 번째는 결정학적 데이터를 이용한 하이브리드 접근법을 적용했다.세 번째는 X선 결정 기술에 의해 결정되는 생쥐 VDAC-1 결정체용이었다.VDAC-1의 3D 구조 3가지 프로젝트는 많은 구조적 특징을 드러냈다.첫째, VDAC-1은 가닥이 홀수인 외부막 β-바렐 단백질의 새로운 구조 등급을 나타낸다.또 다른 측면은 잔류물 E73의 음전하 사이드 체인이 소수성 막 환경을 지향한다는 것이다.세 개의 다른 실험실이 서로 다른 실험 선원에 따라 획득한 19 가닥의 3D 구조는 기본 멤브레인 선원의 전자파 및 AFM 데이터를 적합시키고 VDAC-1의 생물학적으로 관련 있는 상태를 나타낸다.[10]
메커니즘
30mV(양극 또는 음극)를 초과하는 멤브레인 전위에서 VDAC는 폐쇄 상태를 가정하고 전압이 이 임계값 아래로 떨어지면 개방 상태로 전환한다.두 주 모두 단순 염분 통과를 허용하지만 VDAC는 대부분의 대사물이 속하는 유기 음이온으로 훨씬 엄격하다.[13]단백질 내의 순응적 변화에 대한 커플링 전압 변화를 위한 정확한 메커니즘은 아직 해결되지 않았지만, 토마스 외 연구 결과에 따르면 단백질이 닫힌 형태로 전환될 때 전압 변화가 채널에서 단백질의 큰 부분을 제거하고 유효 모공 반경을 감소시킨다고 한다.[14]Glu-152뿐만 아니라 여러 라이신 잔류물이 단백질 내 특히 중요한 센서 잔류물로 관련되어 있다.[15]
생물 함수
전압에 의존하는 이온 채널은 외부 미토콘드리아 막 전체에 걸쳐 신진대사 및 에너지 유량을 조절하는 데 핵심적인 역할을 한다.ATP, ADP, 화농산염, 악성산염 및 기타 대사물의 수송에 관여하고 있으므로 대사 경로에서 효소와 광범위하게 교신한다.[13]미토콘드리아 효소 크레아틴 키나아제는 물론 ATP 의존형 세포질 효소 헥소키나아제, 글루코키나제, 글리세롤키나아제 등이 모두 VDAC에 결합하는 것으로 밝혀졌다.이 제본은 그들을 미토콘드리아에서 방출된 ATP에 근접하게 한다.특히 헥소키나아제의 결합은 산화인산화에 글리코분해를 결합하는 데 핵심적인 역할을 하는 것으로 추정된다.[14]또한 VDAC는 미토콘드리아 내외의 Ca 수송의2+ 중요한 규제 기관이다.Ca는2+ 화농산탈수소효소, 이소시트레이트탈수소효소 등 대사효소의 공동 인자이기 때문에 에너지 생산과 동태 모두 VDAC의 Ca에2+ 대한 투과성에 영향을 받는다.[16]
질병 관련성
VDAC는 또한 세포사멸의 역할을 하는 것으로 나타났다.[17]세포사멸 중 VDAC는 미토콘드리아 투과성 전환 공극을 시토크롬 c와 같은 세포유발 인자의 방출로 수정한다.그러나 VDAC는 미토콘드리아 투과성 전환 모공에서 필수적인 구성요소는 아니다.cyt. c는 미토콘드리온 내 산화 인산화에서 필수적인 역할을 하지만, cytosol에서는 caspases라고 불리는 단백질 분해 효소를 활성화시켜 세포 사망에 큰 역할을 한다.[18]VDAC가 제조한 cyt. c 릴리즈의 메커니즘이 아직 완전히 설명되지는 않았지만, 일부 연구는 개별 서브유닛 사이의 과점화가 cyt. c가 통과할 수 있는 큰 유연한 모공을 만들 수 있다고 제안한다.[19]더 중요한 요인은 cyt c.의 방출도 bcl-2 단백질 계열에 의해 조절된다는 것이다: bax는 VDAC와 직접 상호 작용하여 모공 크기를 증가시키고 cyt. c 릴리즈를 촉진하는 반면, 항 사포토틱 Bcl-xL은 정반대의 효과를 일으킨다.[20]실제로 VDAC를 억제하는 항체가 격리된 미토콘드리아와 전체 세포 모두에서 박스 매개 ct. 방출을 방해하는 것으로 나타났다.[21]이 세포사멸의 핵심 역할은 VDAC가 화학 요법 약물의 잠재적 타겟임을 시사한다.
예
효모는 이 계열의 두 멤버(Gene POR1, PO2)를 포함하고 있으며 척추동물은 적어도 세 멤버(Gene VDAC1, VDAC2, VDAC3)를 가지고 있다.[11]
인간은 대부분의 상위 진핵생물과 마찬가지로 VDAC1, VDAC2, VDAC3의 세 가지 다른 VDAC를 인코딩한다.TOM40 및 TOM40L와 함께 이들은 진화적으로 연관된 β-바렐 계열을 나타낸다.[22]
식물은 가장 많은 수의 VDAC를 가지고 있다. 아라비독시스(Arabidopsis)는 4개의 다른 VDAC를 부호화하지만 이 숫자는 다른 종에서는 더 클 수 있다.[23]
참조
- ^ Hoogenboom BW, Suda K, Engel A, Fotiadis D (2007). "The supramolecular assemblies of voltage-dependent anion channels in the native membrane". J. Mol. Biol. 370 (2): 246–55. doi:10.1016/j.jmb.2007.04.073. PMID 17524423.
- ^ Blachly-Dyson, E; Forte, M (September 2001). "VDAC channels". IUBMB Life. 52 (3–5): 113–8. doi:10.1080/15216540152845902. PMID 11798022. S2CID 38314888.
- ^ Sabirov RZ, Merzlyak PG (June 2012). "Plasmalemmal VDAC controversies and maxi-anion channel puzzle". Biochim. Biophys. Acta. 1818 (6): 1570–80. doi:10.1016/j.bbamem.2011.09.024. PMID 21986486.
- ^ De Pinto, V.; Messina, A.; Lane, D. J. R.; Lawen, A. (2010). "Voltage-dependent anion-selective channel (VDAC) in the plasma membrane". FEBS Letters. 584 (9): 1793–1799. doi:10.1016/j.febslet.2010.02.049. PMID 20184885. S2CID 3391282.
- ^ Niehage, C.; Steenblock, C.; Pursche, T.; Bornhäuser, M.; Corbeil, D.; Hoflack, B. (2011). Borlongan, Cesario V (ed.). "The Cell Surface Proteome of Human Mesenchymal Stromal Cells". PLOS ONE. 6 (5): e20399. Bibcode:2011PLoSO...620399N. doi:10.1371/journal.pone.0020399. PMC 3102717. PMID 21637820.
- ^ Benz R (1994). "Permeation of hydrophilic solutes through mitochondrial outer membranes: review on mitochondrial porins". Biochim. Biophys. Acta. 1197 (2): 167–196. doi:10.1016/0304-4157(94)90004-3. PMID 8031826.
- ^ Mannella CA (1992). "The 'ins' and 'outs' of mitochondrial membrane channels". Trends Biochem. Sci. 17 (8): 315–320. doi:10.1016/0968-0004(92)90444-E. PMID 1384178.
- ^ Dihanich M (1990). "The biogenesis and function of eukaryotic porins". Experientia. 46 (2): 146–153. doi:10.1007/BF02027310. PMID 1689252. S2CID 2199583.
- ^ Forte M, Guy HR, Mannella CA (1987). "Molecular genetics of the VDAC ion channel: structural model and sequence analysis". J. Bioenerg. Biomembr. 19 (4): 341–350. doi:10.1007/BF00768537. PMID 2442148. S2CID 10219032.
- ^ a b Hiller S, Abramson J, Mannella C, Wagner G, Zeth K (September 2010). "The 3D structures of VDAC represent a native conformation". Trends Biochem. Sci. 35 (9): 514–21. doi:10.1016/j.tibs.2010.03.005. PMC 2933295. PMID 20708406.
- ^ a b Sampson MJ, Lovell RS, Davison DB, Craigen WJ (1996). "A novel mouse mitochondrial voltage-dependent anion channel gene localizes to chromosome 8". Genomics. 36 (1): 192–196. doi:10.1006/geno.1996.0445. PMID 8812436.
- ^ Zeth K (2010). "Structure and evolution of mitochondrial outer membrane proteins of beta-barrel topology". Biochim. Biophys. Acta. 1797 (6–7): 1292–9. doi:10.1016/j.bbabio.2010.04.019. PMID 20450883.
- ^ a b Blachly-Dyson, E. & Forte, M. (2001). "VDAC Channels". IUBMB Life. 52 (3–5): 113–18. doi:10.1080/15216540152845902. PMID 11798022. S2CID 38314888.
- ^ a b Colombini M, Blachly-Dyson E, Forte M (1996). "VDAC, a channel in the outer mitochondrial membrane". Ion Channels. 4: 169–202. doi:10.1007/978-1-4899-1775-1_5. ISBN 978-1-4899-1777-5. PMID 8744209.
- ^ Thomas L, Blachly-Dyson E, Colombini M, Forte M (June 1993). "Mapping of residues forming the voltage sensor of the voltage-dependent anion-selective channel". Proc. Natl. Acad. Sci. U.S.A. 90 (12): 5446–9. Bibcode:1993PNAS...90.5446T. doi:10.1073/pnas.90.12.5446. PMC 46737. PMID 7685903.
- ^ Shoshan-Barmatz V; Gincel D. (2003). "The voltage-dependent anion channel: characterization, modulation, and role in mitochondrial function in cell life and death". Cell Biochem. Biophys. 39 (3): 279–92. doi:10.1385/CBB:39:3:279. PMID 14716081. S2CID 33628015.
- ^ Lemasters JJ; Holmuhamedov E. (2006). "Voltage-dependent anion channel (VDAC) as mitochondrial governator--thinking outside the box". Biochim. Biophys. Acta. 1762 (2): 181–90. doi:10.1016/j.bbadis.2005.10.006. PMID 16307870.
- ^ Tsujimoto Y, Shimizu S (2002). "The voltage-dependent anion channel: an essential player in apoptosis". Biochimie. 84 (2–3): 187–93. doi:10.1016/S0300-9084(02)01370-6. PMID 12022949.
- ^ Zalk R; Israelson A; Garty ES; Azoulay-Zohar H; Shoshan-Barmatz V. (2005). "Oligomeric states of the voltage-dependent anion channel and cytochrome c release from mitochondria". Biochem. J. 386 (1): 73–83. doi:10.1042/BJ20041356. PMC 1134768. PMID 15456403.
- ^ Shimizu S; Narita M; Tsujimoto Y. (1999). "Bcl-2 family proteins regulate the release of apoptogenic cytochrome c by the mitochondrial channel VDAC". Nature. 399 (6735): 483–7. Bibcode:1999Natur.399..483S. doi:10.1038/20959. PMID 10365962. S2CID 4423304.
- ^ Shimizu S; Matsuoka Y; Shinohara Y; Yoneda Y; Tsujimoto Y. (2001). "Essential role of voltage-dependent anion channel in various forms of apoptosis in mammalian cells". J. Cell Biol. 152 (2): 237–50. doi:10.1083/jcb.152.2.237. PMC 2199613. PMID 11266442.
- ^ Bay DC, Hafez M, Young MJ, Court DA (June 2012). "Phylogenetic and coevolutionary analysis of the β-barrel protein family comprised of mitochondrial porin (VDAC) and Tom40". Biochim. Biophys. Acta. 1818 (6): 1502–19. doi:10.1016/j.bbamem.2011.11.027. PMID 22178864.
- ^ Homblé F, Krammer E, Prevost M (June 2012). "Plant VDAC: facts and speculations". Biochim. Biophys. Acta. 1818 (6): 1486–501. doi:10.1016/j.bbamem.2011.11.028. PMID 22155681.


